ΒΙΟΠΛΗΡΟΦΟΡΙΚΗ
ΕΡΓΑΣΤΗΡΙΑΚΗ ΑΣΚΗΣΗ
ΒΑΣΕΙΣ ΔΕΔΟΜΕΝΩΝ ΝΟΥΚΛΕΪΝΙΚΩΝ ΟΞΕΩΝ ΚΑΙ ΠΡΩΤΕΪΝΩΝ
Νικόλαος Χ. Παπανδρέου & Σταύρος Ι. Χαμόδρακας
Τομέας Βιολογίας Κυττάρου & Βιοφυσικής
Τμήμα Βιολογίας
Παν/μιο Αθηνών
Μάρτιος 2002
ΒΙΟΠΛΗΡΟΦΟΡΙΚΗ
ΕΡΓΑΣΤΗΡΙΑΚΗ ΑΣΚΗΣΗ
ΒΑΣΕΙΣ ΔΕΔΟΜΕΝΩΝ ΝΟΥΚΛΕΪΝΙΚΩΝ ΟΞΕΩΝ ΚΑΙ ΠΡΩΤΕΪΝΩΝ

Νικόλαος Χ. Παπανδρέου & Σταύρος Ι. Χαμόδρακας
Τομέας Βιολογίας Κυττάρου & Βιοφυσικής
Τμήμα Βιολογίας
Παν/μιο Αθηνών
Μάρτιος 2002
Οι βάσεις δεδομένων ακολουθιών και δομών DNA και πρωτεϊνών αποτελούν το χαρακτηριστικότερο δείγμα της αλματώδους εξέλιξης που έχει επιτελεστεί τα τελευταία χρόνια στη Βιολογία. Όταν πρωτοξεκίνησε η δημιουργία τους ο όγκος της πληροφορίας ήταν τόσο μικρός που και ένας μικρός αριθμός ερευνητών αρκούσε για την συντήρηση και για την ανανέωση των βάσεων αυτών. Αν κάποιος ερευνητής ενδιαφέρονταν να έχει πρόσβαση στις εγγραφές της βάσης επικοινωνούσε με τους επιστημονικούς υπευθύνους και εκείνοι του έστελναν με συμβατικό ταχυδρομείο όλη τη βάση η οποία αρκούσε να αποθηκευτεί ακόμη και σε μερικές δισκέτες ή μια μαγνητοταινία.
Την τελευταία δεκαετία όμως η τεχνολογική εξέλιξη βοήθησε στη διεκπεραίωση μεγάλου όγκου πειραματικής εργασίας η οποία σε συνάρτηση με τον διαρκή προσδιορισμό γονιδιωμάτων διαφόρων οργανισμών αύξησε τον όγκο της πληροφορίας στο επίπεδο της ακολουθίας και όχι μόνο, σε δυσθεώρητα μεγέθη. Οι βάσεις πλέον δεν περιέχουν απλώς πολλά δεδομένα αλλά και η διαδικασία ανανέωσης τους είναι απαραίτητα καθημερινή υπόθεση. Πλέον η συντήρηση μιας βάσης απαιτεί ένα πολυάριθμο επιτελείο επιστημόνων οι οποίοι ασχολούνται αποκλειστικά με το σχολιασμό (annotation) των νεοεισερχόμενων δεδομένων καθώς και με τη διόρθωση λαθών των ήδη υπαρχόντων. Χαρακτηριστικά παραδείγματα αποτελούν η βάση πρωτεϊνικών ακολουθιών SWISS-PROT που περιέχει 104948 ακολουθίες (Rel. 40.9 - Ιανουάριος 2002), ενώ η EMBL Nucleotide Sequence Database που περιέχει νουκλεοτιδικές αλληλουχίες έχει 14366182 εγγραφές (Rel. 69 - Δεκέμβριος 2001). Η πρόσβαση στις βάσεις αυτές είναι πλέον εύκολη μέσω της χρήσης του Διαδικτύου. Ο χρήστης μπορεί να επισκεφτεί την ιστοσελίδα που διατηρείται από τους υπευθύνους της βάσης και να κάνει αναζητήσεις αποθηκεύοντας στον υπολογιστή του δεδομένα του άμεσου ενδιαφέροντός του. Παράλληλα έχουν δημιουργήθεί και μια σειρά από βάσεις που αποσκοπούν στην ταξινόμηση της πληροφορίας στο επίπεδο της ακολουθίας και της δομής προκειμένου να οργανωθεί η πληροφορία και να εξαχθούν συμπεράσματα για την βιολογική τους σημασία.
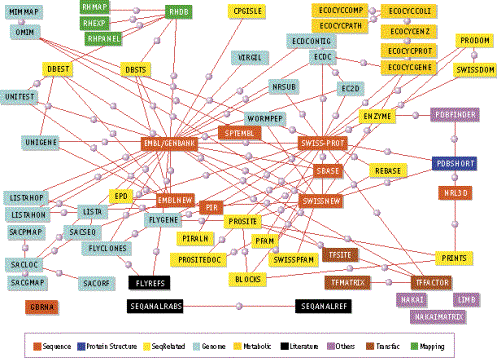
Παρακάτω παρατίθενται οι κυριότερες κατηγορίες βάσεων νουκλεϊνικών οξέων και πρωτεϊνών και οι κυριότεροι αντιπρόσωποι τους.
Α. Βάσεις δεδομένων νουκλεοτιδικών αλληλουχιών.
Οι βάσεις δεδομένων νουκλεοτιδικών αλληλουχιών αποτελούν τις μεγαλύτερες βάσεις στο ευρύτερο πεδίο της Βιολογίας τόσο από άποψη του όγκου της πληροφορίας που περιέχουν όσο και από την άποψη του εκθετικού ρυθμού συσσώρευσης δεδομένων που εμφανίζουν. Τα τελευταία χρόνια λόγω της εξέλιξης της τεχνολογίας στην εύρεση της αλληλουχίας (sequencing) πολυνουκλεοτιδίων έγινε εφικτός σε μικρό χρονικό διάστημα, ο προσδιορισμός της αλληλουχίας ολόκληρων γονιδιωμάτων αρκετών οργανισμών όπως ο άνθρωπος. Σε αρκετές περιπτώσεις μάλιστα υπάρχουν εξειδικευμένες βάσεις δεδομένων που περιέχουν τις αλληλουχίες για ένα και μόνο οργανισμό (π.χ. Flybase a database of the Drosophila genome http://flybase.bio.indiana.edu/ ).
Εδώ πρέπει να σημειώσουμε τις τρεις μεγαλύτερες βάσεις δεδομένων νουκλεοτιδικών αλληλουχιών που είναι ελεύθερα διαθέσιμες στην ακαδημαϊκή κοινότητα. Πρόκειται για τις GENBANK (NCBI), DNA Data Bank of Japan (DDBJ) και EMBL Nucleotide Sequence Database (ΕΒΙ) οι οποίες σε συνεργασία έχουν δημιουργήσει την International Nucleotide Sequence Database Collaboration. Η συνεργασία μεταξύ των βάσεων περιλαμβάνει την ανταλλαγή σε καθημερινή βάση εγγραφών που κατατίθενται ανεξάρτητα σε κάθε βάση δεδομένων έχοντας θέσει παράλληλα και κοινούς κανόνες για την ταξινόμηση και το σχολιασμό των δεδομένων. Στο παρακάτω σχήμα παρουσιάζεται η ροή της πληροφορίας ανάμεσα στις βάσεις.
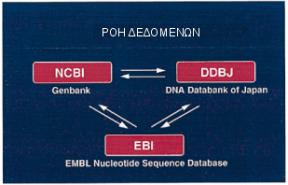
Λίγα λόγια για κάθε βάση δεδομένων που συμμετέχει στην International Nucleotide Sequence Database Collaboration :
GENBANK: Η GENBANK1 ( http://www.ncbi.nlm.nih.gov/Genbank/index.html ) αποτελεί μια βάση δεδομένων νουκλεοτιδικών αλληλουχιών που είναι ελεύθερα διαθέσιμη στην επιστημονική κοινότητα. Βρίσκεται υπό την αιγίδα του Εθνικού Ινστιτούτου Υγείας των Η.Π.Α. Η κύρια πηγή της πληροφορίας που περιέχεται στην GENBANK προέρχεται από απευθείας υποβολές δεδομένων όπως προκύπτουν από πειραματικές διεργασίες διαφόρων ερευνητικών ομάδων. Τα νεοεισερχόμενα δεδομένα υφίστανται επεξεργασία και προστίθενται σχόλια (annotation) για την διευκόλυνση των ερευνητών. Ανά τακτά χρονικά διαστήματα τα ήδη κατατεθειμένα δεδομένα επανεξετάζονται και γίνονται διορθώσεις αν προκύπτουν νέα δεδομένα σχετικά με τις εγγραφές. Η διαδικασία κατάθεσης των δεδομένων μπορεί να πραγματοποιηθεί πολύ γρήγορα μέσω του Διαδικτύου με την συμπλήρωση κατάλληλης φόρμας και στη συνέχεια οι υπεύθυνοι της βάσης αναλαμβάνουν το σχολιασμό της εγγραφής και τη δημοσιοποίηση της στην βάση. Έχει διαπιστωθεί ότι κάθε 14 μήνες ο αριθμός των νουκλεοτιδικών βάσεων που περιέχονται στην GENBANK διπλασιάζεται με αποτέλεσμα η παρούσα έκδοση (Rel. 128, Φεβρουάριος 2002) να περιέχει 15465325 ακολουθίες με τον συνολικό αριθμό βάσεων να φτάνει τις 17089143893.
EMBL-Bank: Η EMBL Nucleotide Sequence Database2 ( http://www.ebi.ac.uk/embl/ ) αποτελεί τη μεγαλύτερη βάση νουκλεοτιδικών αλληλουχιών στην Ευρώπη και βρίσκεται υπό την αιγίδα του Ευρωπαϊκού Εργαστηρίου Μοριακής Βιολογίας (EMBL). Εδράζεται και συντηρείται στο Ευρωπαϊκό Ινστιτούτο Βιοπληροφορικής (EBI) στο Cambridge, UK. Τα δεδομένα προέρχονται από ανεξάρτητα ερευνητικά εργαστήρια καθώς και από ομάδες που ασχολούνται με τον προσδιορισμό των γονιδιωμάτων διαφόρων οργανισμών. Η κατάθεση ακολουθιών στην EMBL-Bank είναι μια διαδικασία απλή και πραγματοποιείται μέσω του Διαδικτύου κατ' αντίστοιχο τρόπο με αυτό της GENBANK. Στη συνέχεια οι νεοεισερχόμενες ακολουθίες υφίστανται επεξεργασία και σχολιασμό από τους υπευθύνους της βάσης προτού γίνουν διαθέσιμες στην επιστημονική κοινότητα. Επιπλέον μέσω του Διαδικτύου παρέχονται μια σειρά από εργαλεία ανάλυσης ακολουθιών (π.χ. Fasta, BLAST). Η τελευταία έκδοση της EMBL-Bank (Rel. 69 - Μάρτιος 2002) περιέχει 15960527 εγγραφές ενώ ο συνολικός αριθμός των νουκλεοτιδίων φτάνει τα 17868806247.
DDJB: H DNA Databank of Japan (DDJB - http://www.ddbj.nig.ac.jp/ ) ιδρύθηκε το 1986 στο Εθνικό Ινστιτούτο Γενετικής (NIG) το οποίο βρίσκεται υπό την αιγίδα του Υπουργείου Παιδείας, Επιστημών και Αθλητισμού της Ιαπωνίας. Αποτελεί τη μοναδική διεθνώς αναγνωρισμένη βάση νουκλεοτιδικών αλληλουχιών στην Ιαπωνία ενώ η κύρια πηγή δεδομένων της είναι οι εργασίες Ιαπώνων ερευνητών. Επιπλέον παρέχονται μια σειρά από εργαλεία για την ανάλυση των νουκλεοτιδικών αλληλουχιών. Η παρούσα έκδοση της DDJB (Rel. 48, Ιανουάριος 2002) περιέχει 15016100 εγγραφές ενώ ο συνολικός αριθμός των νουκλεοτιδικών βάσεων που περιέχονται στις ακολουθίες είναι 16197713855.
Β. Βάσεις δεδομένων πρωτεϊνικών ακολουθιών και βάσεις για την ανάλυση ακολουθιών.
Η SWISS-PROT3 ( http://www.expasy.ch/sprot/ ), είναι μια βάση δεδομένων πρωτεϊνικών ακολουθιών που ιδρύθηκε το 1986 και στις μέρες μας συντηρείται από το Ελβετικό ινστιτούτο Βιοπληροφορικής (Swiss Institute of Bioinformatics) σε συνεργασία με το Ευρωπαϊκό Ινστιτούτο Βιοπληροφορικής (European Bioinformatics Institute). H παρούσα έκδοση της SWISS-PROT περιέχει 104948 καταχωρήσεις (Ιανουάριος 2002) στις οποίες εκτός από την ακολουθία να υπάρχουν και συμπληρωματικά σχόλια όπως, βιβλιογραφικές αναφορές, γενικά στοιχεία δευτεροταγούς δομής, σύνδεσμοι σε άλλες βάσεις δεδομένων σχετικές με κάθε εγγραφή καθώς και σημειώσεις για τη βιολογική λειτουργία (αν είναι γνωστές) και άλλες χρήσιμες πληροφορίες.
Η Protein Information Resource4 (PIR - http://pir.georgetown.edu/ ) εδράζεται στο Πανεπιστήμιο του Georgetown και αποτελεί τμήμα του Εθνικού Ιδρύματος Βιοϊατρικής Έρευνας (NBRF) των Η.Π.Α. Η PIR περιλαμβάνει μια σειρά από βάσεις δεδομένων που σχετίζονται με τη μελέτη των πρωτεϊνών με κυριότερη από αυτές την PIR-International Protein Sequence Database (PSD). H PSD αποτελεί όπως και η SWISS-PROT μια βάση δεδομένων πρωτεϊνικών ακολουθιών συνοδευόμενη από συμπληρωματικά σχόλια. Τα δεδομένα της PSD προκύπτουν από την συνεργασία της PIR με τo Munich Information Center for Protein Sequences (MIPS) και την Japanese International Protein Information Database (JIPID). H τελευταία έκδοση της PSD (Rel. 71.03, Φεβραυάριος 2002) περιλαμβάνει 283138 εγγραφές. Πρέπει να σημειωθεί ότι η PIR-PSD σε 4 υποενότητες τις PIR1, PIR2, PIR3 και PIR4. Μεταξύ των PIR1 και PIR2 δεν υπάρχει ουσιαστική διαφορά. Ο διαχωρισμός διατηρείται κυρίως για ιστορικούς λόγους. Οι ενότητες αυτές περιέχουν το 99% των εγγραφών της βάσης και τα κριτήρια ταξινόμησης και σχολιασμού είναι ακριβώς τα ίδια. Αντίθετα οι εγγραφές της PIR3 δεν έχουν ακόμη υποστεί έλεγχο και σχολιασμό. Τα περιεχόμενα της PIR4 είναι ακολουθίες οι οποίες είτε δεν συναντώνται στη φύση είτε δεν εκφράζονται υπό φυσιολογικές συνθήκες. Επίσης μπορεί να περιέχει ακολουθίες που έχουν συντεθεί de novo σε εργαστήριο. Σε κάθε περίπτωση πάντως έχουν υποστεί έλεγχο και σχολιασμό από τους υπεύθυνους της βάσης.
Η PROSITE5 ( http://www.expasy.ch/prosite/ ) είναι μια βάση ταξινόμησης σε οικογένειες πρωτεϊνικών ακολουθιών και αυτοτελών περιοχών ακολουθιών (sequence domains). Βασίζεται στη γενικότερη παρατήρηση ότι ενώ υπάρχει ένας τεράστιος αριθμός διαφορετικών πρωτεϊνών στη φύση, αυτές μπορούν να ομαδοποιηθούν με βάση την ομοιότητα στην ακολουθία τους σε ένα μικρό αριθμό οικογενειών. Οι πρωτεΐνες ή οι αυτοτελείς δομικές περιοχές που ανήκουν στην ίδια οικογένεια έχουν την ίδια λειτουργία και προέρχονται από κοινό πρόγονο. Είναι φανερό ότι πρωτεΐνες που ανήκουν στην ίδια οικογένεια, έχουν τμήματα της ακολουθίας τους που είναι περισσότερο συντηρημένα στην πορεία της εξέλιξης τους. Αυτές οι περιοχές σχετίζονται άμεσα με τη λειτουργία τους και με τη δομή των πρωτεϊνών στο χώρο. Αναλύοντας τις ακολουθίες πρωτεϊνών που ανήκουν στην ίδια οικογένεια είναι δυνατό να προκύψει ένα 'αποτύπωμα' χαρακτηριστικό για κάθε ομάδα, ικανό ώστε να τη διαχωρίζει από τις άλλες πρωτεϊνικές αλληλουχίες που δεν ανήκουν στην οικογένεια αυτή. Μια ανάλογη περίπτωση αποτελεί η λήψη αποτυπωμάτων από την αστυνομία. Ένα αποτύπωμα είναι ικανό για να ταυτοποιήσει ένα άτομο. Παρόμοια και στις πρωτεΐνες η χρήση ενός τέτοιου αποτυπώματος μπορεί να χρησιμεύσει για να ταξινομηθεί μια άγνωστη πρωτεϊνική αλληλουχία σε μια γνωστή οικογένεια πρωτεϊνών δινόντας μας ενδείξεις για την πιθανή λειτουργία τους. Αυτή τη στιγμή η PROSITE περιέχει 'αποτυπώματα' για 1000 περίπου οικογένειες. Για κάθε οικογένεια υπάρχει λεπτομερής ανάλυση για τη δομή και τη λειτουργία των πρωτεϊνών αυτών.
Γ. Βάσεις δεδομένων δομικής βιολογίας.
Protein Data Bank: H Protein Data Bank6 (PDB - www.rcsb.org ) αποτελεί τη μοναδική βάση παγκοσμίως όπου είναι κατατεθειμένες οι τρισδιάστατες δομές βιολογικών μακρομορίων. Ιδρύθηκε το 1971 στα Brookhaven National Laboratories (BNL) των ΗΠΑ και περιελάμβανε 7 δομές μακρομορίων όπως αυτές προέκυψαν από κρυσταλλογραφικές μελέτες. Ο ρυθμός αύξησης των εγγραφών στη δεκαετία του '70 ήταν πολύ μικρός. Από το 1980 και μετά λόγω της τεχνολογικής εξέλιξης σε κάθε στάδιο του προσδιορισμού δομών ο ρυθμός προσθήκης δεδομένων στην PDB αυξήθηκε δραματικά. Πλέον στην βάση περιλαμβάνονται και δομές όπως προκύπτουν με φασματοσκοπία Πυρηνικού Μαγνητικού Συντονισμού (NMR). Στην παρούσα φάση (Μάρτιος 2002) η PDB περιλαμβάνει 17493 δομές βιομορίων. Οι εγγραφές στην PDB εκτός από τις συντεταγμένες των ατόμων που απαρτίζουν τη δομή περιλαμβάνουν και επιπρόσθετα βοηθητικά στοιχεία όπως βιβλιογραφικές αναφορές, λεπτομέρειες για τον προσδιορισμό της δομής καθώς και άλλα στοιχεία που προκύπτουν από τη συγκεκριμένη δομή. Κάθε δομή προτού διατεθεί στο κοινό υφίσταται έλεγχο για την ορθότητα της με τη χρήση ειδικού λογισμικού. Στη συνέχεια εφόσον περάσει τις δοκιμές με επιτυχία αποκτά ένα χαρακτηριστικό κωδικό και προστίθεται στη βάση.
CATH: Η CATH7 ( http://www.biochem.ucl.ac.uk/bsm/cath_new/index.html ) είναι μια βάση ιεραρχικής ταξινόμησης των πρωτεϊνικών δομών που είναι κατατεθειμένες στην PDB με βάση τις αυτοτελείς δομικές περιοχές (domains) που τις απαρτίζουν. Για τον καταρτισμό της CATH δεν λαμβάνονται υπόψιν μη πρωτεϊνικές δομές ενώ οι πρωτεϊνικές δομές που περιέχονται πρέπει να είναι προσδιορισμένες σε διακριτικότητα υψηλότερη των 3 Angstroms. Η CATH χρησιμοποιεί κυρίως αυτοματοποιημένες μεθόδους για την ταξινόμηση. Σε ειδικές περιπτώσεις όμως τα ανθρώπινα κριτήρια είναι δυνατόν να δώσουν καλύτερα αποτελέσματα από τις αυτοματοποιημένες μεθόδους όποτε και προτιμούνται.
Τα 4 κύρια επίπεδα της ιεραρχίας είναι η Τάξη (Class), η Αρχιτεκτονική (Architecture), η Τοπολογία (Οικογένεια διπλώματος) (Topology (fold family)) και η Ομόλογη Οικογένεια (Homologous superfamily).
Οι πρωτεΐνες που αποτελούνται από περισσότερα του ενός domains αναλύονται στα επιμέρους στοιχεία αυτόματα με βάση ειδικούς αλγόριθμους αναγνώρισης domains. Με την αυτόματη διαδικασία κατατάσσονται το 53% των δομών. Οι υπόλοιπες διαχωρίζονται στα επιμέρους domains με παρατηρήσεις που προκύπτουν είτε από τους αλγόριθμους αυτόματου διαχωρισμού είτε από τη βιβλιογραφία. Η ταξινόμηση πραγματοποιείται μόνο στις αυτοτελείς δομικές περιοχές.
Ιεραρχία στην CATH
C - Τάξη (Class): Η κατάταξη σε τάξεις πραγματοποιείται λαμβάνοντας υπόψιν τα στοιχεία δευτεροταγούς δομής μιας αυτοτελούς δομικής περιοχής. Αυτό γίνεται αυτόματα για το 90% των πρωτεϊνών ενώ για τις υπόλοιπες χρησιμοποιούνται κυρίως δεδομένα από τη βιβλιογραφία. Με βάση την κατάταξη προκύπτουν 4 μεγάλες ομάδες: mainly-alpha (Τα στοιχεία δευτεροταγούς τους δομής είναι στην συντριπτική τους πλειοψηφία α-έλικες), mainly-beta (κυρίως β-εκτεταμένες δομές) και alpha-beta (εναλλασσόμενες α/β και α+β δομές) και δομές με χαμηλό ποσοστό δευτεροταγών δομών.
A - Αρχιτεκτονική (Architecture): Η ταξινόμηση πραγματοποιείται με βάση την γενικότερη δομή της αυτοτελούς δομικής περιοχής (domain), με βάση τον προσανατολισμό των στοιχείων δευτεροταγούς δομής μη λαμβάνοντας υπόψιν όμως τον τρόπο διασύνδεσης μεταξύ τους π.χ. βαρέλια (barrels).
Τ - Τοπολογία (Topology): Σε αυτό το επίπεδο οι δομές ταξινομούνται με βάση τον προσανατολισμό των στοιχείων δευτεροταγούς δομής αλλά και με βάση την σύνδεση αυτών των στοιχείων μεταξύ τους.
Η - Ομόλογη οικογένεια (Homology superfamily): Ομαδοποίηση των δομικών στοιχείων που εμφανίζουν 35% ομοιότητα μεταξύ τους στο επίπεδο της αλληλουχίας τους με αποτέλεσμα να θεωρείται ότι προέρχονται από ένα κοινό πρόγονο.
S - Αλληλουχία (Sequence family): Τα μέλη της εμφανίζουν ομοιότητα στο επίπεδο της ακολουθίας πάνω από 35% με αποτέλεσμα να θεωρείται ότι έχουν παρόμοια δομή και λειτουργία.
SCOP: Η βάση SCOP8 ( http://scop.mrc-lmb.cam.ac.uk/scop/index.html ) έχει σαν βασικό στόχο την ανάλυση των δομικών και εξελικτικών σχέσεων μεταξύ όλων των πρωτεϊνών γνωστής δομής κατατεθειμένων στην Protein Data Bank (PDB). Για την αναγνώριση των παραπάνω σχέσεων και την ταξινόμηση με βάση τις σχέσεις αυτές, των πρωτεϊνών η διαδικασία δεν είναι αυτοματοποιημένη αλλά πραγματοποιείται αποκλειστικά με βάση τον ανθρώπινο παράγοντα μετά από λεπτομερή μελέτη και σύγκριση των πρωτεϊνικών δομών. Αυτοματοποιημένες μέθοδοι χρησιμοποιούνται μόνο για την ομοιογένεια των δεδομένων που περιέχονται στη βάση.
Η ταξινόμηση των πρωτεϊνών όπως αναφέρθηκε παραπάνω γίνεται με βάση δομικές και εξελικτικές σχέσεις. Τα βασικά επίπεδα ταξινόμησης είναι η οικογένεια (Family), υπερ-οικογένεια (Superfamily), το δίπλωμα (Fold) και η τάξη (Class).
Οικογένεια (Family): Ξεκάθαρη εξελιγκτική σχέση μεταξύ των μελών.
Οι πρωτεΐνες που ταξινομούνται σε μια οικογένεια έχουν ξεκάθαρη εξελικτική σχέση μεταξύ τους. Η ομοιότητα σε επίπεδο ακολουθίας είναι της τάξης του 30% και άνω. Υπάρχουν όμως περιπτώσεις όπου οι δομές και η λειτουργία είναι παρόμοιες υποδηλώνοντας κοινό πρόγονο ενώ η ομοιότητα σε επίπεδο ακολουθίας να είναι μικρότερη του 30% (σφαιρίνες, 15%).
Υπερ-οικογένεια (Superfamily): Τα μέλη της έχουν πιθανά προέλθει από κοινό πρόγονο.
Στο επίπεδο της υπερ-οικογένειας κατατάσσονται πρωτεΐνες που εμφανίζουν πολύ μικρή ομοιότητα στο επίπεδο της ακολουθίας αλλά τα δομικά τους χαρακτηριστικά και η λειτουργία τους υποδηλώνουν πιθανή κοινή προέλευση.
Δίπλωμα (Fold): Εμφάνιση ομοιότητας σε επίπεδο δομής.
Οι πρωτεΐνες που εμφανίζουν το ίδιο δίπλωμα έχουν τα ίδια σε μεγάλο βαθμό χαρακτηριστικά δευτεροταγούς δομής, με κοινό προσανατολισμό και τις ίδιες τοπολογικές συνδέσεις μεταξύ τους. Πρωτεΐνες που έχουν το ίδιο δίπλωμα αλλά δεν είναι όμοιες από άποψη αμινοξικής ακολουθίας έχουν ορισμένα περιφερειακά στοιχεία της δευτεροταγούς τους δομής και στροφές ανόμοια και όσον αφορά στο μέγεθος και όσον αφορά στη διαμόρφωση. Πρωτεΐνες που εμφανίζουν κοινό δίπλωμα δεν είναι απαραίτητο να έχουν κοινή εξελικτική προέλευση.
Τάξη (Class): Τέσσερις κύριες δομικές κατηγορίες πρωτεϊνών έχουν ταυτοποιηθεί με βάση το δίπλωμα των στοιχείων δευτεροταγούς δομής τους, τις αll-α (η δομή σχηματίζεται από α-έλικες), all-β (η δομή αποτελείται από β-πτυχωτές επιφάνειες), α/β (α-έλικες και β-πτυχωτές επιφάνειες εναλλάσσονται στην δομή της πρωτεΐνης) και α+β (α-έλικες και β-πτυχωτές επιφάνειες βρίσκονται σε διακριτές περιοχές της δομής).
Δ. Ολοκληρωμένα συστήματα ανάκτησης πληροφοριών από βάσεις δεδομένων.
Το SRS είναι ένα ισχυρό, εύχρηστο σύστημα διαχείρισης δεδομένων το οποίο διατίθεται από την εταιρία LION Bioscience. Το SRS μέσω ενός φιλικού προς το χρήστη γραφικού περιβάλλοντος δίνει την δυνατότητα αναζήτησης και ανάκτησης δεδομένων από περισσότερες από 400 βάσεις δεδομένων οι οποίες μπορεί να είναι αποθηκευμένες στον ίδιο κεντρικό υπολογιστή. Το μεγάλο του πλεονέκτημα είναι ότι μπορείς να κάνεις ταυτόχρονη αναζήτηση για ένα ζήτημα άμεσου ενδιαφέροντος σε παραπάνω από μια βάσεις δεδομένων που δεν περιέχουν ανάλογου είδους πληροφορία και η μορφοποίηση των δεδομένων σε καθεμιά να είναι διαφορετική. Ένα άλλο μεγάλης σημασίας πλεονέκτημα είναι η ταχύτητα με την οποία εκτελούνται οι αναζητήσεις παρά το γεγονός ότι διαχειρίζεται πραγματικά τεράστιο όγκο πληροφορίας λόγω του μεγάλου αριθμού βάσεων που μπορεί να διαχειρίζεται ταυτόχρονα. Τέλος δίνεται η δυνατότητα στον κάτοχο του συστήματος να ενσωματώνει σε αυτό και βάσεις που έχει δημιουργήσει ο ίδιος ή προγράμματα για κάθε είδος υπολογιστική ανάλυση χωρίς να επηρεάζεται η απόδοση του συστήματος.
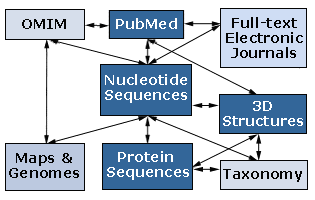
Το Entrez αποτελεί ένα σύστημα διαχείρισης για την αναζήτηση και ανάκτηση πληροφοριών ανάλογο του SRS όλων των βάσεων δεδομένων που περιέχονται στο NCBI (National Center for Biotechnology Information) των ΗΠΑ. Το Entrez δίνει τη δυνατότητα αναζήτησης σε βάσεις δεδομένων νουκλεοτιδικών και πρωτεϊνικών ακολουθιών, δομές βιομορίων, γονιδιωμάτων και στη βάση για την αναζήτηση βιβλιογραφίας MEDLINE μέσω του ίδιου γραφικού περιβάλλοντος επιτρέποντας και πιο πολύπλοκες αναζητήσεις ανάμεσα στα στοιχεία τους. Βέβαια το γεγονός ότι περιορίζεται μόνο στις βάσεις δεδομένων του NCBI και ότι δεν επιτρέπει πολύπλοκες αναζητήσεις το καθιστούν υποδεέστερο έναντι του SRS.
Παρακάτω παρατίθεται ένα σχεδιάγραμμα των αλληλεπιδράσεων μεταξύ των βάσεων που διαχειρίζεται το σύστημα Entrez.
1. D. A. Benson et al., Nucl. Acids Res., (2000), 28:15-18.
2. G. Stoesser et al., Nucl. Acids Res., (2002), 30:21-26.
3. A. Bairoch, R. Apweiler, Nucleic Acids Res., (2000), 28:45-48.
4. C. H. Wu et .al., Nucleic Acids Res., (2002), 30:35-37
5. K. Hofmann et al., Nucleic Acids Res., (1999), 27:215-219.
6. H. M. Berman et al., Nucl. Acids Res., (2000), 28:235-242.
7. F. Pearl et al., Nucl. Acids Res., (2001), 29:223-227.
8. A. G. Murzin et al., J. Mol. Biol., (1995), 247:536-540.
Α. Καταρχήν θα μελετήσετε μια εγγραφή στην βάση δεδομένων νουκλεοτιδικών ακολουθιών GENBANK.
Τα βήματα που ακολουθούμε είναι τα εξής:
1. Αρχικά, στο φυλλομετρητή ιστού που διαθέτουμε (Internet Explorer, Netscape, κλπ) πληκτρολογούμε τη διεύθυνση:
http://www.ncbi.nlm.nih.gov/Genbank/index.html
2. Στη συνέχεια πληκτρολογήστε στο κενό πεδίο του προγράμματος SEARCH GENBANK τη φράση κλειδί dihydrofolate reductase. Πρόκειται για το ένζυμο Διϋδροφολική Αναγωγάση (DHFR) που συμμετέχει στο βιοχημικό μονοπάτι σύνθεσης της Θυμίνης. Ακολούθως πατήστε το κουμπί Go.
3. Στη σελίδα των αποτελεσμάτων επιλέξτε μια από τις εγγραφές και προσπαθήστε με τη βοήθεια του ΠΑΡΑΡΤΗΜΑΤΟΣ να αναγνωρίσετε τα πεδία της εγγραφής.
Β. Στη συνέχεια της άσκησης θα έχουμε πρόσβαση στις βάσεις δεδομένων SWISS-PROT και PIR, από τις οποίες θα έχουμε την ευκαιρία να αντλήσουμε κάποιες πρωτεϊνικές ακολουθίες και να μελετήσουμε την οργάνωση συγκεκριμένων εγγραφών.
1. Στο φυλλομετρητή ιστού πληκτρολογούμε τη διεύθυνση:
http://www.sanger.ac.uk/srs6bin/cgi-bin/wgetz?-page+top+-newId
που μας οδηγεί στο SRS του Sanger Institute ενός ερευνητικού κέντρου που ασχολείται με την ανάλυση γονιδιωμάτων και χρηματοδοτείται από την εταιρία Wellcome Trust και την Βρετανική Κυβέρνηση. Αφού ξεκινήσουμε την εφαρμογή πατώντας το κουμπί START εμφανίζεται στην οθόνη μας ένας κατάλογος επιλογής των βάσεων δεδομένων με τις οποίες επιθυμούμε να εργαστούμε.
* Η πρώτη εργασία που έχετε να κάνετε είναι να διαπιστώσετε σε τι είδους βάσεις δεδομένων μας δίνει τη δυνατότητα το SRS να κάνουμε αναζήτηση δεδομένων. Πρέπει να σημειώσουμε ότι κάθε φορέας που διαθέτει το σύστημα SRS μπορεί να παρέχει πρόσβαση σε διαφορετικές βάσεις σε σχέση με τους υπόλοιπους φορείς.
2. Επιλέγουμε τη SwissProt (πατώντας στο αντίστοιχο κουτάκι) που περιέχει πρωτεϊνικές ακολουθίες και χρήσιμα σχόλια, και συνεχίζουμε (panel Query Forms, κουμπί 'Standard').
3. Τώρα μπορούμε να πραγματοποιήσουμε την αναζήτησή μας, τροφοδοτώντας τις περιοχές κειμένου που μας παρέχονται με τα στοιχεία των πρωτεϊνών που μας ενδιαφέρουν.
Συγκεκριμένα, κάθε ομάδα θα πραγματοποιήσει αναζήτηση με λέξη κλειδί DIHYDROFOLATE σε όλο το κείμενο (All Text) των καταχωρήσεων της SwissProt. Για να χωρέσουν τα αποτελέσματα της αναζήτησής σας σε μια σελίδα επιλέξτε να έχετε προβολή ανά 200 καταχωρήσεις (Number of entries to display per page: 200).
4. Πατώντας Submit Query αρχίζει η αναζήτηση.
5. Τα αποτελέσματα της αναζήτησης στην SWISS-PROT εμφανίζονται υπό μορφή πίνακα 4 στηλών της μορφής:

Επιλέγοντας ένα σύνδεσμο από την αριστερή στήλη οδηγείστε στα περιεχόμενα της εγγραφής της SWISS-PROT.
Κάθε ομάδα θα επιλέξει μια από τις εγγραφές και θα προσπαθήσει να αναγνωρίσει τα πεδία μιας εγγραφής SWISS-PROT με τη βοήθεια του ΠΑΡΑΡΤΗΜΑΤΟΣ.
**Επαναλαμβάνοντας την ίδια ακριβώς διαδικασία δοκιμάστε να μελετήσετε εγγραφές της βάσης PIR-PSD για την ίδια πρωτεΐνη (Διϋδροφολική Αναγωγάση) επιλέγοντας αυτή την φορά αναζήτηση με την ίδια λέξη κλειδί στην PIR αντί της SWISS-PROT.
Γ. Για την αναζήτηση patterns σε πρωτεϊνικές ακολουθίες θα χρησιμοποιήσουμε την βάση PROSITE. Για την αναζήτηση στην PROSITE θα χρησιμοποιήσουμε πάλι το σύστημα SRS. Τα βήματα που ακολουθούμε είναι:
1. Οδηγούμαστε στην κεντρική σελίδα του SRS ακολουθώντας την ίδια διαδικασία όπως στις αναζητήσεις μας στην PIR και την SWISS-PROT.
2. Επιλέγουμε την PROSITE από την ενότητα SeqRelated και συνεχίζουμε (panel Query Forms, κουμπί 'Standard').
3. Τώρα μπορούμε να πραγματοποιήσουμε την αναζήτησή μας, τροφοδοτώντας τις περιοχές κειμένου που μας παρέχονται με τα στοιχεία των πρωτεϊνών που μας ενδιαφέρουν. Συγκεκριμένα, κάθε ομάδα θα πραγματοποιήσει αναζήτηση με λέξη κλειδί DHFR σε όλο το κείμενο (All Text) των καταχωρήσεων της PROSITE. Για να χωρέσουν τα αποτελέσματα της αναζήτησής σας σε μια σελίδα επιλέξτε να έχετε προβολή ανά 200 καταχωρήσεις (Number of entries to display per page: 200).
4. Πατώντας Submit Query αρχίζει η αναζήτηση.
5. Επιλέξτε το αποτέλεσμα της αναζήτησης και προσπαθήσετε να αναγνωρίσετε όλα τα πεδία της εγγραφής καθώς και να ερμηνεύσετε το pattern που λαμβάνει χώρα σε αυτή την οικογένεια των πρωτεϊνικών ακολουθιών.
Δ. Τέλος θα έχουμε πρόσβαση στη Βάση Δεδομένων Πρωτεϊνικών Δομών (PDB) και θα μελέτησουμε πως ταξινομούνται πρωτεϊνικές δομές στην CATH και την SCOP. Η πρόσβασή μας στις καταχωρήσεις της είναι δυνατή με τη βοήθεια του προγράμματος SRS, όμως θα έχουμε απευθείας πρόσβαση μέσω του link που βρίσκεται στην Αρχική Σελίδα του εργαστηρίου Βιοφυσικής και Βιοπληροφορικής του Τομέα Βιολογίας ( ενότητα 'Tertiary Structure Analysis', υποενότητα 'Databanks').
Επιλέγουμε το link που μας οδηγεί στην Αρχική Σελίδα της PDB.
Αρχίζουμε την αναζήτησή μας με το πρόγραμμα Search επίλέγοντας το σύνδεσμο SearchFields.
Στο πεδίο Compound Information πληκτρολογήστε DIHYDROFOLATE.
Στο Result Display Options επιλέγουμε All προκειμένου στην σελίδα εμφάνισης των αποτελεσμάτων να εμφανιστούν όλες οι εγγραφές.
Πατάμε το κουμπί SEARCH προκειμένου να ξεκινήσει η αναζήτηση.
Τα αποτελέσματα της αναζήτησης εμφανίζονται στο φυλλομετρητή μας δίνοντας μας όμως ελάχιστα στοιχεία για κάθε εγγραφή. Προκειμένου να δούμε λεπτομέρειες μιας εγγραφής επιλέξτε το EXPLORE σε κάποια από αυτές της λίστας.
Στη συνέχεια από το Menu DownLoad/Display File θα επιλέξετε να έχετε πρόσβαση στα πλήρη δεδομένα με τις ατομικές συντεταγμένες (complete with coordinates σε PDB format και μορφή απλού ASCII κειμένου).
Προσπαθήστε να αναγνωρίσετε τις βασικές εγγραφές της καταχώρησης με τη βοήθεια του ΠΑΡΑΡΤΗΜΑΤΟΣ.
Επιλέξτε στην συνέχεια το σύνδεσμο (link) Structural Neighbors.
Η σελίδα που εμφανίζεται εμφανίζει διασυνδέσεις για τις βάσεις CATH και SCOP.
Επιλέξτε κάθε μια από αυτές προκειμένου να διαπιστώσετε πως είναι ταξινομημένη η εγγραφή που μελετήσατε στις βάσεις CATH και SCOP.
1. Εγγραφή της GENBANK για το γονίδιο της Διϋδροφολικής Αναγωγάσης (DHFR) από τον οργανισμό Lactobacillus casei.
LOCUS LBADHFR 1145 bp DNA linear BCT 26-APR-1993
DEFINITION Lactobacillus casei dihydrofolate reductase gene (DHFR), complete cds.
ACCESSION M10922
VERSION M10922.1 GI:149539
KEYWORDS dihydrofolate reductase; methotrexate-resistant.
SOURCE L.casei (methotrexate-resistant (MTX-R) strain) DNA, clone pWDLcB1.
ORGANISM Lactobacillus casei
Bacteria; Firmicutes; Bacillus/Clostridium group; Lactobacillales;
Lactobacillaceae; Lactobacillus.
REFERENCE 1 (bases 1 to 1145)
AUTHORS Andrews,J., Clore,G.M., Davies,R.W., Gronenborn,A.M.,
Gronenborn,B., Kalderon,D., Papadopoulos,P.C., Schaefer,S.,
Sims,P.F.G. and Stancombe,R.
TITLE Nucleotide sequence of the dihydrofolate reductase gene of
methotrexate-resistant Lactobacillus casei
JOURNAL Gene 35, 217-222 (1985)
MEDLINE 85286353
COMMENT A -35 and a -10 region are located at positions 134-139 and
158-163 respectively. An RBS is found at 306-312.
FEATURES Location/Qualifiers
source 1..1145
/organism="Lactobacillus casei"
/db_xref="taxon:1582"
mRNA 174..866
/note="DHFR mRNA (5' and 3' ends +/- 2 bp)"
CDS 321..812
/note="dihydrofolate reductase"
/codon_start=1
/transl_table=11
/protein_id="AAA25237.1"
/db_xref="GI:149540"
/translation="MTAFLWAQDRDGLIGKDGHLPWHLPDDLHYFRAQTVGKIMVVGR
RTYESFPKRPLPERTNVVLTHQEDYQAQGAVVVHDVAAVFAYAKQHPDQELVIAGGAQ
IFTAFKDDVDTLLVTRLAGSFEGDTKMIPLNWDDFTKVSSRTVEDTNPALTHTYEVWQ
KKA"
BASE COUNT 320 a 243 c 290 g 292 t
ORIGIN 1 bp upstream of EcoRI site.
1 gaattcattc atacattcgg cgatgcgcat ctttacgtca atcatcttga ccaaattaaa
61 gagcagctca gtcgcacgcc gcggccggca ccgactttac agttgaatcc ggataaacat
121 gatattttcg actttgacat gaaggatatt aagttgctta attacgatcc ttatccggcc
181 attaaggcac cggttgccgt ttaatcgcta gaagacggca agtcataaca agtgtctgat
241 tgctttgtca ggtttaccaa tgacacaaaa ggcgccattt tgttcggctt tggattgcat
301 actcaaagga ggggtctcga atgaccgcat ttttatgggc acaggatcgc gatggcttaa
361 ttggcaaaga tggtcatttg ccatggcatt taccggatga tttacattat ttccgggcgc
421 agacagttgg taagatcatg gtcgttggtc ggcgcaccta tgaaagtttt cctaaacgtc
481 ctttacctga gcgaaccaat gttgttttga cccatcagga agactatcaa gcgcaaggtg
541 ccgtggtcgt gcatgatgtt gcggcggttt ttgcttatgc taagcagcat cccgatcagg
601 aactggtcat tgctggcggt gcacagatct ttacggcttt taaagatgat gtcgatacgt
661 tactggtaac acgtttggct ggcagttttg aaggcgatac gaaaatgatt ccattaaact
721 gggatgattt taccaaagtc tccagccgca ccgttgaaga taccaatccg gcgctgacgc
781 acacttatga ggtttggcaa aagaaggctt aagcagaagc cgatgaccgg aattggtggt
841 tgccagctgg tgcgggtgtg agtttagacg catatttgcg tgcatttaaa aaatcgtctc
901 tcgtattatc tggcaaaaca aaaaccgcag tccgctgcat caaaaacaaa ctcagccgcg
961 ggcaagccaa aagcaccggc aaaaaaacgg cgccaaaaca gaagtcaaag ttgacatatg
1021 ctgagcagat agagtatgat aagctccaac aagaacttga tgaattagac gagcagttgg
1081 ccaaggttaa agcagaaatg gcgcaggtca atggtgagga ttacgtgaag ctgggcgatc
1141 tgcag
//
Επεξηγήσεις των σημαντικότερων πεδίων μιας εγγραφής στην GENBANK
LOCUS: Περιέχει ένα μικρό όνομα για τον χαρακτηρισμό της εγγραφής.
DEFINITION: Μια λεπτομερής περιγραφή της ακολουθίας.
ACCESSION: Κωδικός που αποκτά μια νεοεισερχόμενη εγγραφή χαρακτηριστικός για την GENBANK. O κωδικός παραμένει σταθερός
VERSION: Ειδικός κωδικός που απαρτίζεται από το πρωταρχικό Accession Number, ακολουθεί το σύμβολο της τελείας και στη συνέχεια ένας αριθμός που δηλώνει την έκδοση της παρούσας εγγραφής.
KEYWORDS: Χαρακτηριστικές λέξεις-κλειδιά που σχετίζονται με την νουκλεοτιδική αλληλουχία και τις ιδιότητες των προϊόντων της.
SOURCE: Βιολογική πηγή της ακολουθίας όπου αναφέρεται ο οργανισμός από των οποίο έχει απομονωθεί με τα ιδιαίτερα χαρακτηριστικά του (πιθανές μεταλλάξεις, πλασμίδια κ.α.).
ORGANISM: Οργανισμός απ' όπου προήλθε η ακολουθία. Ακολουθείται η διώνυμη ονομασία κατά Λινναίο. Επίσης παρατίθεται και η συστηματική ταξινόμηση του οργανισμού.
- Τα παρακάτω πεδία σχετίζονται με την δημοσιευμένη εργασία στην οποία αναφέρεται ο προσδιορισμός της παρούσας ακολουθίας.
REFERENCE: Περιέχει τον αριθμό της αναφοράς καθώς και το μήκος της ακολουθίας που έχει προσδιοριστεί στην παρούσα εργασία.
AUTHORS: Αναφέρονται οι συμμετέχοντες στην διεξαγωγή της παρούσας εργασίας.
TITLE: Τίτλος της δημοσιευμένης εργασίας.
JOURNAL: Περιέχει λεπτομέρειακα στοιχεία για την αναζήτηση της αναφοράς όπως είναι ο τίτλος του περιοδικού που εκδόθηκε, τεύχος, ημερομηνία έκδοσης και σελίδες που καταλαμβάνει στο συγκεκριμένο τεύχος.
MEDLINE: Κωδικός για την βιβλιογραφική αναφορά στην βάση δεδομένων MEDLINE.
COMMENT: Περιέχει κάποιες γενικές παρατηρήσεις, ή αναφορές και σε άλλες βάσεις.
FEATURES: Πίνακας που περιέχει πληροφορίες σχετικά με τα προϊόντα της ακολουθίας όπως πολυπεπτιδικές αλυσίδες (από μετάφραση) και RNA (από μεταγραφή) και στοιχεία από πειραματικά δεδομένα που καταδεικνύουν τη βιολογική της σημασία.
BASE COUNT: Αριθμητική ανάλυση της ακολουθίας στα επιμέρους συστατικά της. Περιέχει το σύνολο καταλοίπων Αδενίνης, Γουανίνης, Κυτοσίνης, Θυμίνης.
ORIGIN: Θέση της πρώτης βάσης της κατατεθειμένης ακολουθίας σε σχέση με το γονιδίωμα από το οποίο έχει απομονωθεί.
Ακριβώς από κάτω παρατίθεται η ακολουθία της παρούσας εγγραφής.
Η αναπαράσταση της ακολουθίας είναι της μορφής:
1 gatctggtgg ccatggcggg agcaaatcag ccgatcccat cccgaactcg gccgtcaaat
61 gccccagcgc ccatgatact ctgcctcaag gcacggaaaa gtcggtcgcc gccaga
//
---------+---------+---------+---------+---------+---------+---------+---------
1 10 20 30 40 50 60 70 79
- Τα νουκλεοτίδια απεικονίζονται με τον κώδικα ενός γράμματος ανάλογα με την αζωτούχο βάση την οποία αποτελούνται.
- Κάθε ακολουθία αποτελείται από 60 αμινοξικά κατάλοιπα ανά γραμμή, σε ομάδες των δέκα αμινοξικών καταλοίπων, ξεκινώντας πάντα από την θέση 11 της γραμμής. Οι ομάδες των 10 καταλοίπων χωρίζονται μεταξύ τους με κενό διάστημα.
- Από τη θέση 9 της γραμμής και προς τα αριστερά υπάρχει ένας αριθμός που δείχνει την αρίθμηση του πρώτου καταλοίπου κάθε γραμμής.
//: Λήξη της εγγραφής.
2. Εγγραφή της SWISS-PROT για την πρωτεϊνική ακολουθία της Διϋδροφολικής Αναγωγάσης (DHFR) από τον οργανισμό Lactobacillus casei.
ID DYR_LACCA STANDARD; PRT; 162 AA.
AC P00381;
DT 21-JUL-1986 (Rel. 01, Created)
DT 30-MAY-2000 (Rel. 39, Last sequence update)
DT 16-OCT-2001 (Rel. 40, Last annotation update)
DE Dihydrofolate reductase (EC 1.5.1.3).
GN FOLA OR DHFR.
OS Lactobacillus casei.
OC Bacteria; Firmicutes; Bacillus/Clostridium group; Lactobacillaceae;
OC Lactobacillus.
OX NCBI_TaxID=1582;
RN [1]
RP SEQUENCE FROM N.A.
RX MEDLINE=85286353; PubMed=3928445; [NCBI, ExPASy, EBI, Israel, Japan]
RA Andrews J., Clore G.M., Davies R.W., Gronenborn A.M., Gronenborn B.,
RA Kalderon D., Papadopoulos P.C., Schaefer S., Sims P.F.G.,
RA Stancombe R.;
RT "Nucleotide sequence of the dihydrofolate reductase gene of
RT methotrexate-resistant Lactobacillus casei.";
RL Gene 35:217-222(1985).
RN [2]
RP SEQUENCE.
RX MEDLINE=78242349; PubMed=98527; [NCBI, ExPASy, EBI, Israel, Japan]
RA Freisheim J.H., Bitar K.G., Reddy A.V., Blankenship D.T.;
RT "Dihydrofolate reductase from amethopterin-resistant Lactobacillus
RT casei. Sequences of the cyanogen bromide peptides and complete
RT sequences of the enzyme.";
RL J. Biol. Chem. 253:6437-6444(1978).
RN [3]
RP SEQUENCE OF 1-51.
RX MEDLINE=77181453; PubMed=405008; [NCBI, ExPASy, EBI, Israel, Japan]
RA Batley K.E., Morris H.R.;
RT "Dihydrofolate reductase from Lactobacillus casei: N-terminal
RT sequence and comparison with the substrate binding region of other
RT reductases.";
RL Biochem. Biophys. Res. Commun. 75:1010-1014(1977).
RN [4]
RP X-RAY CRYSTALLOGRAPHY (1.7 ANGSTROMS).
RX MEDLINE=83056868; PubMed=6815179; [NCBI, ExPASy, EBI, Israel, Japan]
RA Filman D.J., Bolin J.T., Matthews D.A., Kraut J.;
RT "Crystal structures of Escherichia coli and Lactobacillus casei
RT dihydrofolate reductase refined at 1.7-A resolution. II. Environment
RT of bound NADPH and implications for catalysis.";
RL J. Biol. Chem. 257:13663-13672(1982).
RN [5]
RP STRUCTURE BY NMR.
RX MEDLINE=91283478; PubMed=1905571; [NCBI, ExPASy, EBI, Israel, Japan]
RA Carr M.D., Birdsall B., Frenkiel T.A., Bauer C.J., Jimenez-Barbero J.,
RA Polshakov V.I., McCormick J.E., Roberts G.C.K., Feeney J.;
RT "Dihydrofolate reductase: sequential resonance assignments using 2D
RT and 3D NMR and secondary structure determination in solution.";
RL Biochemistry 30:6330-6341(1991).
RN [6]
RP STRUCTURE BY NMR.
RX MEDLINE=96018856; PubMed=7547901; [NCBI, ExPASy, EBI, Israel, Japan]
RA Morgan W.D., Birdsall B., Polshakov V.I., Sali D., Kompis I.,
RA Feeney J.;
RT "Solution structure of a brodimoprim analogue in its complex with
RT Lactobacillus casei dihydrofolate reductase.";
RL Biochemistry 34:11690-11702(1995).
RN [7]
RP STRUCTURE BY NMR.
RX MEDLINE=98181015; PubMed=9514736; [NCBI, ExPASy, EBI, Israel, Japan]
RA Gargaro A.R., Soteriou A., Frenkiel T.A., Bauer C.J., Birdsall B.,
RA Polshakov V.I., Barsukov I.L., Roberts G.C.K., Feeney J.;
RT "The solution structure of the complex of Lactobacillus casei
RT dihydrofolate reductase with methotrexate.";
RL J. Mol. Biol. 277:119-134(1998).
RN [8]
RP STRUCTURE BY NMR.
RX MEDLINE=99190070; PubMed=10091649; [NCBI, ExPASy, EBI, Israel, Japan]
RA Polshakov V.I., Birdsall B., Frenkiel T.A., Gargaro A.R., Feeney J.;
RT "Structure and dynamics in solution of the complex of Lactobacillus
RT casei dihydrofolate reductase with the new lipophilic antifolate drug
RT trimetrexate.";
RL Protein Sci. 8:467-481(1999).
CC -!- CATALYTIC ACTIVITY: 5,6,7,8-tetrahydrofolate + NADP(+) = 7,8-
CC dihydrofolate + NADPH.
CC -!- PATHWAY: ESSENTIAL STEP FOR DE NOVO GLYCINE AND PURINE SYNTHESIS,
CC DNA PRECURSOR SYNTHESIS, AND FOR THE CONVERSION OF DUMP TO DTMP.
CC -!- SUBUNIT: MONOMER.
CC -!- MISCELLANEOUS: THIS BACTERIAL STRAIN IS RESISTANT TO THE FOLIC
CC ACID ANALOG METHOTREXATE (AMETHOPTERIN).
CC -!- SIMILARITY: BELONGS TO THE DIHYDROFOLATE REDUCTASE FAMILY.
CC --------------------------------------------------------------------------
CC This SWISS-PROT entry is copyright. It is produced through a collaboration
CC between the Swiss Institute of Bioinformatics and the EMBL outstation -
CC the European Bioinformatics Institute. There are no restrictions on its
CC use by non-profit institutions as long as its content is in no way
CC modified and this statement is not removed. Usage by and for commercial
CC entities requires a license agreement (See http://www.isb-sib.ch/announce/
CC or send an email to license@isb-sib.ch).
CC --------------------------------------------------------------------------
DR EMBL; M10922; AAA25237.1; -. [EMBL / GenBank / DDBJ] [CoDingSequence]
DR PIR; A00394; RDLBD.
DR PIR; A12987; A12987.
DR PIR; A24036; A24036.
DR PDB; 3DFR; 31-JAN-84. [ExPASy / RCSB]
DR PDB; 1DIS; 14-NOV-95. [ExPASy / RCSB]
DR PDB; 1DIU; 14-NOV-95. [ExPASy / RCSB]
DR PDB; 1AO8; 25-FEB-98. [ExPASy / RCSB]
DR PDB; 1BZF; 18-MAY-99. [ExPASy / RCSB]
DR InterPro; IPR001796; DHFR.
DR InterPro; Graphical view of domain structure.
DR Pfam; PF00186; DiHfolate_red; 1.
DR PRINTS; PR00070; DHFR.
DR PROSITE; PS00075; DHFR; 1.
DR ProDom [Domain structure / List of seq. sharing at least 1 domain]
DR BLOCKS; P00381.
DR DOMO; P00381.
DR PROTOMAP; P00381.
DR PRESAGE; P00381.
DR DIP; P00381.
DR ModBase; P00381.
DR SWISS-2DPAGE; GET REGION ON 2D PAGE.
KW Oxidoreductase; NADP; Methotrexate resistance; 3D-structure;
KW One-carbon metabolism.
FT INIT_MET 0 0
FT ACT_SITE 21 21 CHEMICAL STUDIES SUGGEST THAT IT
FT PARTICIPATES IN ENZYME FUNCTION.
FT ACT_SITE 26 26 INVOLVED IN BINDING METHOTREXATE.
FT ACT_SITE 31 31 INVOLVED IN BINDING METHOTREXATE.
FT ACT_SITE 43 43 MAY BE INVOLVED IN BINDING THE COENZYME
FT NADPH.
FT ACT_SITE 57 57 INVOLVED IN BINDING METHOTREXATE.
FT CONFLICT 8 8 D -> N (IN REF. 2).
FT CONFLICT 90 90 P -> L (IN REF. 2).
FT STRAND 2 7
FT TURN 9 10
FT STRAND 12 15
FT TURN 16 17
FT STRAND 18 18
FT HELIX 24 32
FT TURN 33 36
FT STRAND 38 42
FT HELIX 43 48
FT TURN 55 56
FT STRAND 58 62
FT TURN 66 67
FT TURN 71 72
FT STRAND 74 76
FT HELIX 79 88
FT STRAND 94 96
FT HELIX 100 105
FT TURN 106 106
FT HELIX 107 109
FT STRAND 112 118
FT STRAND 126 127
FT HELIX 133 135
FT STRAND 136 144
FT HELIX 149 151
FT STRAND 153 160
SQ SEQUENCE 162 AA; 18308 MW; 4484539182675921 CRC64;
TAFLWAQDRD GLIGKDGHLP WHLPDDLHYF RAQTVGKIMV VGRRTYESFP KRPLPERTNV
VLTHQEDYQA QGAVVVHDVA AVFAYAKQHP DQELVIAGGA QIFTAFKDDV DTLLVTRLAG
SFEGDTKMIP LNWDDFTKVS SRTVEDTNPA LTHTYEVWQK KA
//
Επεξηγήσεις των σημαντικότερων πεδίων μιας εγγραφής SWISS-PROT
ID (Identification):
Είναι της μορφής Entry_name data_class; molecule_type; sequence length
Entry_name: Το όνομα της ακολουθίας χαρακτηριστικό για τη βάση SWISS-PROT.
π.χ. DYR_LACCA. Το πρώτο τμήμα υποδηλώνει το όνομα της αλληλουχίας όπως είναι κατατεθειμένο στην βάση. Μπορεί να έχει μήκος μέχρι 4 χαρακτήρες. Το δεύτερο καθορίζει το είδος από το οποίο προέρχεται η αλληλουχία. Μπορεί να έχει μήκος μέχρι 5 χαρακτήρες.
data_class: Δηλώνει αν η εγγραφή έχει σχολιαστεί ή όχι με βάση τα κριτήρια της βάσης SWISS-PROT.
molecule_type: Δηλώνει σε ποια ομάδα μακρομορίων ανήκει η ακολουθία. Για τις εγγραφές της SWISS-PROT είναι PRT (Protein).
sequence length: To μήκος της ακολουθίας σε αμινοξικά κατάλοιπα (ΑΑ).
AC (Accession number): Είναι ένας χαρακτηριστικός κωδικός που αποκτά μια πολυπεπτιδική αλυσίδα όταν κατατίθεται στην βάση. Χρησιμεύει στην αναγνώριση εγγραφών ανάμεσα στις διαφορετικές εκδόσεις της βάσης όπως αυτή ανανεώνεται ανά τακτά χρονικά διαστήματα.
DT (Date): Αναγραφή ημερομηνίας για τη δημιουργία της παρούσας εγγραφής, τελευταίας τροποποιήσης, προσθήκης σχολίων.
DE (Description): Γενική περιγραφή για την ακολουθία.
GN (Gene name): Γονίδιο από το οποίο με μετάφραση προέκυψε η αμινοξική ακολουθία.
OS (Organism Species): Οργανισμός απ' όπου προήλθε η ακολουθία. Ακολουθείται η διώνυμη ονομασία κατά Λινναίο.
OG (Organelle): Επεξηγεί αν το γονίδιο που κωδικοποιεί την συγκεκριμένη αλληλουχία εδράζεται σε μιτοχόνδρια, χλωροπλάστες ή πλασμίδιο.
OC (Organism Classification): Συστηματική ταξινόμηση του οργανισμού απ'όπου προήλθε η ακολουθία.
ΟΧ (Organism taxonomy cross-reference): Παραπομπή σε βάση δεδομένων συστηματικής ταξινόμησης των οργανισμών.
RN (Reference number): Αύξων αριθμός αναφοράς σχετικής με την παρούσα εγγραφή.
RP (Reference Position): Περιέχει λίγες πληροφορίες σχετικές με το τι πραγματεύεται η συγκεκριμένη αναφορά.
RX (Reference cross-reference): Παραπομπές σε βιβλιογραφικές βάσεις δεδομένων π.χ. PUBMED.
RA (Reference author): Λίστα με τους συγγραφείς της παρούσας αναφοράς.
RT (Reference title): Τίτλος της παρούσας εργασίας όπως δημοσιεύτηκε σε επιστημονικά περιοδικά.
RL (Reference Location): Περιοδικό ή βιβλίο όπου δημοσιεύτηκε η παρούσα εργασία.
CC (Comments): Το πεδίο αυτό περιέχει μία σειρά από πληροφορίες πάσης φύσεως σχετικές με την ακολουθία. Χωρίζεται σε υπο-πεδία όπως:
CATALYTIC ACTIVITY: Περιγραφή της αντίδρασης που καταλύεται αν η ακολουθία είναι ένζυμο.
ALTERNATIVE PRODUCTS: Αναφέρεται αν υπάρχουν σχετικές με αυτή αλληλουχίες που έχουν προκύψει από εναλλακτικό μάτισμα.
FUNCTION: Σύντομη περιγραφή της λειτουργίας που συμμετέχει η ακολουθία.
SUBCELLULAR LOCATION: Θέση της ακολουθίας στο κύτταρο.
SUBUNIT: Το πεδίο εμφανίζεται στην περίπτωση που η ακολουθία συμμετέχει στην δημιουργία τεταρτοταγούς δομής μιας πρωτεΐνης.
Πρέπει να σημειωθεί πως τα παραπάνω είναι μερικά από τα υπο-πεδία που μπορεί να περιέχονται στο πεδίο CC (Comments).
DR (Database cross-reference): To πεδίο αυτό δίνει διασυνδέσεις σε άλλες βάσεις δεδομένων που σχετίζονται με την παρούσα εγγραφή όπως η PDB, η EMBL κ.α. με τους αντίστοιχους κωδικούς τους.
KW (Keyword): Το πεδίο αυτό περιέχει ειδικές λέξεις-κλειδιά για τον χαρακτηρισμό της αλληλουχίας όπως αυτές ταξινομούνται με βάση κριτήρια όπως η λειτουργία και η δομή τους.
FT (Feature Table): Το πεδίο αυτό περιέχει στοιχεία χαρακτηριστικά για την ακολουθία αυτή καθεαυτή και αφορά συγκεκριμένα τμήματά της. Περιλαμβάνει πληροφορίες για:
α. Μεταμεταφραστικές τροποποιήσεις
β. Ποια τμήματα της ακολουθίας είναι υπεύθυνα για την δέσμευση κάποιου μορίου (π.χ. Receptor-Ligand).
γ. Ποια τμήματα της ακολουθίας συμμετέχουν για το σχηματισμό του ενεργού κέντρου αν πρόκειται για ένζυμο.
δ. Στοιχεία για τη δευτεροταγή δομή της αλληλουχίας.
ε. Επίσης μπορεί στο πεδίο αυτό μπορεί και να σημειώνονται και διαφορές στην αλληλουχία εάν έχουν προκύψει και αναφέρονται σε άλλες βιβλιογραφικές αναφορές.
SQ (Sequence): Το πεδίο αυτό περιέχει το μήκος της ακολουθίας σε αμινοξέα (ΑΑ), το μοριακό βάρος (MW) σε Daltons.
Ακολουθεί η αναπαράσταση της ακολουθίας ακολουθώντας τους παρακάτω κανόνες:
- Κάθε αμινοξικό κατάλοιπο απεικονίζεται με τον κώδικα του ενός γράμματος κατά IUPAC.
- Κάθε ακολουθία αποτελείται από 60 αμινοξικά κατάλοιπα ανά γραμμή, σε ομάδες των δέκα αμινοξικών καταλοίπων, ξεκινώντας πάντα από την θέση 6 της γραμμής. Οι ομάδες των 10 καταλοίπων χωρίζονται μεταξύ τους με κενό διάστημα.
//: Τα σύμβολα αυτά υποδηλώνουν το τέλος της εγγραφής.
Π.χ.
SQ SEQUENCE 162 AA; 18308 MW; 4484539182675921 CRC64;
TAFLWAQDRD GLIGKDGHLP WHLPDDLHYF RAQTVGKIMV VGRRTYESFP KRPLPERTNV
VLTHQEDYQA QGAVVVHDVA AVFAYAKQHP DQELVIAGGA QIFTAFKDDV DTLLVTRLAG
SFEGDTKMIP LNWDDFTKVS SRTVEDTNPA LTHTYEVWQK KA
//
3. Εγγραφή της PIR-PSD για την πρωτεϊνική ακολουθία της Διϋδροφολικής Αναγωγάσης (DHFR) από τον οργανισμό Lactobacillus casei.
ENTRY RDLBD #type complete iProClass View of RDLBD
TITLE dihydrofolate reductase (EC 1.5.1.3) [validated] -
Lactobacillus casei
ORGANISM #formal_name Lactobacillus casei
#cross-references taxon:1582
DATE 31-May-1979 #sequence_revision 17-Sep-1997 #text_change
15-Sep-2000
ACCESSIONS A24036; A00394; A12987
REFERENCE A24036
#authors Andrews, J.; Clore, G.M.; Davies, R.W.; Gronenborn, A.M.;
Gronenborn, B.; Kalderon, D.; Papadopoulos, P.C.; Schafer,
S.; Sims, P.F.G.; Stancombe, R.
#journal Gene (1985) 35:217-222
#title Nucleotide sequence of the dihydrofolate reductase gene of
methotrexate-resistant Lactobacillus casei.
#cross-references MUID:85286353
#accession A24036
##molecule_type DNA
##residues 1-163 ##label AND
##cross-references GB:M10922; NID:g149539; PIDN:AAA25237.1;
PID:g149540
##experimental_source clone pWDLcB1
##note sequence from a methotrexate-resistant strain
REFERENCE A00394
#authors Freisheim, J.H.; Bitar, K.G.; Reddy, A.V.; Blankenship,
D.T.
#journal J. Biol. Chem. (1978) 253:6437-6444
#title Dihydrofolate reductase from amethopterin-resistant
Lactobacillus casei. Sequences of the cyanogen bromide
peptides and complete sequence of the enzyme.
#cross-references MUID:78242349
#accession A00394
##molecule_type protein
##residues 2-8,'N',10-90,'L',92-163 ##label FRE
##note sequence from a strain resistant to the folic acid analog
methotrexate (amethopterin); it is not clear whether the
differences reflect strain variations
REFERENCE A12987
#authors Batley, K.E.; Morris, H.R.
#journal Biochem. Biophys. Res. Commun. (1977) 75:1010-1014
#title Dihydrofolate reductase from Lactobacillus casei:
N-terminal sequence and comparison with the substrate
binding region of other reductases.
#cross-references MUID:77181453
#accession A12987
##molecule_type protein
##residues 2-52 ##label BAT
##note sequence from a methotrexate-resistant strain
REFERENCE A50583
#authors Filman, D.J.; Matthews, D.A.; Bolin, J.T.; Kraut, J.
#submission submitted to the Brookhaven Protein Data Bank, June 1982
#cross-references PDB:3DFR
#contents annotation; X-ray crystallography, 1.7 angstroms, 2-8,'N',
10,'N',12-90,'L',92-163
#note dichloromethotrexate-resistant strain
#note strain methotrexate-resistant, expressed in Escherichia
coli
REFERENCE A65422
#authors Morgan, W.D.; Birdsall, B.; Polshakov, V.I.; Sali, D.;
Kompis, I.; Feeney, J.
#submission submitted to the Brookhaven Protein Data Bank, August 1995
#cross-references PDB:1DIS
#contents annotation; conformation by NMR, residues 2-163
REFERENCE A58587
#authors Morgan, W.D.; Birdsall, B.; Polshakov, V.I.; Sali, D.;
Kompis, I.; Feeney, J.
#journal Biochemistry (1995) 34:11690-11702
#title Solution structure of a brodimoprim analogue in its
complex with Lactobacillus casei dihydrofolate reductase.
#cross-references MUID:96018856
#contents annotation; conformation by (1)H-NMR
FUNCTION
#description catalyzes the reduction of dihydrofolic acid to
tetrahydrofolic acid with NADPH; oxidoreductase
#pathway tetrahydrofolate synthesis
CLASSIFICATION SF000194
#superfamily type I dihydrofolate reductase; type I dihydrofolate
reductase homology
KEYWORDS NADP; oxidoreductase
FEATURE
2-163 #product dihydrofolate reductase #status
experimental #label MAT\
2-108 #domain type I dihydrofolate reductase homology
#label DFR\
27,31,58 #binding_site substrate (Asp, Phe, Arg) #status
predicted
SUMMARY #length 163 #molecular_weight 18439
SEQUENCE
5 10 15 20 25 30
1 M T A F L W A Q D R D G L I G K D G H L P W H L P D D L H Y
31 F R A Q T V G K I M V V G R R T Y E S F P K R P L P E R T N
61 V V L T H Q E D Y Q A Q G A V V V H D V A A V F A Y A K Q H
91 P D Q E L V I A G G A Q I F T A F K D D V D T L L V T R L A
121 G S F E G D T K M I P L N W D D F T K V S S R T V E D T N P
151 A L T H T Y E V W Q K K A
Επεξηγήσεις των σημαντικότερων πεδίων μιας εγγραφής PIR-PSD
ENTRY: Κωδικός της ακολουθίας χαρακτηριστικός για την PIR-PSD.
TITLE: Σύντομη περιγραφή της ακολουθίας.
ORGANISM: Οργανισμός απ' όπου προήλθε η ακολουθία. Ακολουθείται η διώνυμη ονομασία κατά Λινναίο.
DATE: Αναγραφή ημερομηνίας για τη δημιουργία της παρούσας εγγραφής, τελευταίας τροποποιήσης, προσθήκης σχολίων.
ACCESSIONS: Είναι ένας χαρακτηριστικός κωδικός που αποκτά μια πολυπεπτιδική αλυσίδα όταν κατατίθεται στην βάση. Χρησιμεύει στην αναγνώριση εγγραφών ανάμεσα στις διαφορετικές εκδόσεις της βάσης όπως αυτή ανανεώνεται ανά τακτά χρονικά διαστήματα.
REFERENCE: Στοιχεία για τις βιβλιογραφικές αναφορές που σχετίζονται με την παρούσα εγγραφή.
FUNCTION: Σύντομη περιγραφή της λειτουργίας που συμμετέχει η ακολουθία.
CLASSIFICATION: Σύνδεσμος που οδηγεί σε μια λίστα από ακολουθίες που ανήκουν στην ίδια οικογένεια όπως κατατάσσονται με βάση την iProClass.
KEYWORDS: Το πεδίο αυτό περιέχει ειδικές λέξεις-κλειδιά για τον χαρακτηρισμό της αλληλουχίας όπως αυτές ταξινομούνται με βάση κριτήρια όπως η λειτουργία και η δομή τους. Επίσης αν επιλεχθούν
FEATURE: Καταγραφή των ιδιαίτερων χαρακτηριστικών της ακολουθίας όπως είναι κατάλοιπα για την πρόσδεση μικρών μορίων.
SUMMARY: Περιέχει στοιχεία για το μοριακό βάρος και το μήκος σε αμινοξικά κατάλοιπα της ακολουθίας.
SEQUENCE: η αλληλουχία των αμινοξέων της πολυπεπτιδικής αλυσίδας η οποία ακολουθεί ειδική μορφοποίηση της μορφής:
- Τα αμινοξικά κατάλοιπα αναπαριστώνται με τον κώδικα του ενός γράμματος κατά IUPAC.
- Η ακολουθία περιλαμβάνει 30 αμινοξικά κατάλοιπα ανά γραμμή τα οποία χωρίζονται μεταξύ τους με κενό διάστημα, ξεκινώντας από τη θέση 7 κάθε γραμμής.
- Από τη θέση 5 της γραμμής και προς τα αριστερά υπάρχει ένας αριθμός που δείχνει την αρίθμηση του πρώτου καταλοίπου κάθε γραμμής.
Π.χ.
5 10 15 20 25 30
1 M T A F L W A Q D R D G L I G K D G H L P W H L P D D L H Y
31 F R A Q T V G K I M V V G R R T Y E S F P K R P L P E R T N
61 V V L T H Q E D Y Q A Q G A V V V H D V A A V F A Y A K Q H
4. Εγγραφή της PROSITE για την πρωτεϊνική ακολουθία της Διϋδροφολικής Αναγωγάσης (DHFR).
ID DHFR; PATTERN. AC PS00075; DT APR-1990 (CREATED); NOV-1997 (DATA UPDATE); JUL-1998 (INFO UPDATE). DE Dihydrofolate reductase signature. PA [LVAGC]-[LIF]-G-x(4)-[LIVMF]-P-W-x(4,5)-[DE]-x(3)-[FYIV]-x(3)-[STIQ]. NR /RELEASE=40.7,103373; NR /TOTAL=69(69); /POSITIVE=68(68); /UNKNOWN=0(0); /FALSE_POS=1(1); NR /FALSE_NEG=4; /PARTIAL=0; CC /TAXO-RANGE=ABEPV; /MAX-REPEAT=1; DR Q05762, DRT1_ARATH, T; Q05763, DRT2_ARATH, T; P45350, DRTS_DAUCA, T; DR P16126, DRTS_LEIAM, T; P07382, DRTS_LEIMA, T; O81395, DRTS_MAIZE, T; DR Q27828, DRTS_PARTE, T; Q27713, DRTS_PLABA, T; P20712, DRTS_PLACH, T; DR P13922, DRTS_PLAFK, T; O02604, DRTS_PLAVI, T; P46103, DRTS_PLAVN, T; DR P51820, DRTS_SOYBN, T; Q07422, DRTS_TOXGO, T; Q27783, DRTS_TRYBB, T; DR Q27793, DRTS_TRYCR, T; P00382, DYR1_ECOLI, T; P12833, DYR3_SALTY, T; DR P11731, DYR5_ECOLI, T; P95524, DYR6_PROMI, T; P27422, DYR7_ECOLI, T; DR Q57452, DYR8_ECOLI, T; Q59397, DYR9_ECOLI, T; P28019, DYR_AEDAL , T; DR P11045, DYR_BACSU , T; P00376, DYR_BOVIN , T; P04382, DYR_BPT4 , T; DR P57243, DYR_BUCAI , T; Q93341, DYR_CAEEL , T; P22906, DYR_CANAL , T; DR P00378, DYR_CHICK , T; P31073, DYR_CITFR , T; Q07801, DYR_CRYNE , T; DR P17719, DYR_DROME , T; P00379, DYR_ECOLI , T; P31074, DYR_ENTAE , T; DR P00380, DYR_ENTFC , T; P43791, DYR_HAEIN , T; P15093, DYR_HALVO , T; DR P27421, DYR_HSVS7 , T; P09503, DYR_HSVSA , T; P22573, DYR_HSVSC , T; DR P00374, DYR_HUMAN , T; P27498, DYR_KLEAE , T; P00381, DYR_LACCA , T; DR Q59487, DYR_LACLA , T; P04753, DYR_MESAU , T; P00375, DYR_MOUSE , T; DR P47470, DYR_MYCGE , T; Q9CBW1, DYR_MYCLE , T; P78028, DYR_MYCPN , T; DR Q98Q32, DYR_MYCPU , T; O33305, DYR_MYCTU , T; P04174, DYR_NEIGO , T; DR Q9JSQ9, DYR_NEIMA , T; Q9K168, DYR_NEIMB , T; P00377, DYR_PIG , T; DR P16184, DYR_PNECA , T; P36591, DYR_SCHPO , T; Q59908, DYR_STAEP , T; DR Q54277, DYR_STAHA , T; Q54801, DYR_STRPN , T; P07807, DYR_YEAST , T; DR Q04515, DYRA_ECOLI, T; P13955, DYRA_STAAU, T; P10167, DYRB_STAAU, T; DR Q59408, DYRC_ECOLI, T; P78218, DYRF_ECOLI, T; DR Q23695, DRTS_CRIFA, N; O62583, DYR_ENCCU , N; Q60034, DYR_THEMA , N; DR Q9PR30, DYR_UREPA , N; DR Q10821, YT00_MYCTU, F; 3D 1AI9; 1AOE; 1DR1; 1DR2; 1DR3; 1DR4; 1DR5; 1DR6; 1DR7; 8DFR; 1DDR; 1DDS; 3D 1DRA; 1DRE; 1DRH; 1DYH; 1DYI; 1DYJ; 1JOL; 1JOM; 1RA1; 1RA2; 1RA3; 1RA8; 3D 1RA9; 1RB2; 1RB3; 1RC4; 1RD7; 1RE7; 1RF7; 1RG7; 1RH3; 1RX1; 1RX2; 1RX3; 3D 1RX4; 1RX5; 1RX6; 1RX7; 1RX8; 1RX9; 1TDR; 3DRC; 4DFR; 5DFR; 6DFR; 7DFR; 3D 1VDR; 1BOZ; 1DHF; 1DLR; 1DRF; 1HFP; 1HFQ; 1HFR; 1OHJ; 1OHK; 2DHF; 1AO8; 3D 1BZF; 1DIS; 1DIU; 3DFR; 1CD2; 1DAJ; 1DYR; DO PDOC00072; //
Επεξηγήσεις των σημαντικότερων πεδίων μιας εγγραφής στην PROSITE
ID (Identification): Είναι της γενικής μορφής
ID ENTRY_NAME; ENTRY_TYPE
Το πρώτο τμήμα είναι η χαρακτηριστική ονομασία που εμφανίζει η εγγραφή χαρακτηριστική για τη βάση PROSITE, ενώ το δεύτερο τμήμα υποδηλώνει τον τύπο της εγγραφής.
AC (ACcession number): Πρόκειται για τον χαρακτηριστικό κωδικό που αποκτά μια νεοεισερχόμενη εγγραφή στην PROSITE και χρησιμεύει στην αναγνώριση της εγγραφής ανάμεσα στις διαφορετικές εκδόσεις της βάσης PROSITE.
DT (DaTe): Το πεδίο αυτό περιέχει τις ημερομηνίες δημιουργίας και τελευταίας ανανέωσης (σχολιασμός) της εγγραφής.
DE (DEscription): Περιέχει μια γενική περιγραφή για την συγκεκριμένη εγγραφή.
PA (PAttern): Στο πεδίο αυτό αναγράφεται το πρότυπο της ακολουθίας (pattern) που ακολουθούν τα μέλη της συγκεκριμένης εγγραφής.
Οι συμβάσεις που ακολουθούμε για την αναπαράσταση του pattern είναι:
NR (Numerical Results): Τα πεδία αυτά περιέχουν στοιχεία που προκύπτουν από την σάρωση (pattern scan) της βάσης SWISS-PROT με το pattern της PROSITE.
Πιο συγκεκριμένα περιλαμβάνουν:
/RELEASE: Η έκδοση της SWISS-PROT που έχει χρησιμοποιηθεί καθώς και ο αριθμός των εγγραφών που περιέχονται σε αυτή.
/TOTAL: Συνολικός αριθμός εγγραφών της SWISS-PROT όπου φαίνεται να συναντάται το pattern.
/POSITIVE: Αριθμός των εγγραφών που είναι βέβαιο ότι συναντάται το pattern και ανήκουν σε οικογένεια της PROSITE.
/UNKNOWN: Αριθμός των εγγραφών που πιθανά ανήκει στην οικογένεια της PROSITE.
/FALSE_POS: Εγγραφές της SWISS-PROT όπου εμφανίζεται το pattern αλλά δεν σχετίζονται με την συγκεκριμένη οικογένεια.
/FALSE_NEG: Αριθμός εγγραφών της SWISS-PROT που ανήκουν στη συγκεκριμένη οικογένεια αλλά δεν βρέθηκαν κατά το pattern scan.
/PARTIAL: Αριθμός ακολουθιών της SWISS-PROT που δεν είναι πλήρεις (fragments), ανήκουν στην συγκεκριμένη οικογένεια της PROSITE, αλλά δεν ανιχνεύονται από τo PROSITE λόγω έλλειψης τμημάτων της ακολουθίας.
CC (Comments): Στα υπο-πεδία του Comments περιέχονται γενικά σχόλια που σχετίζονται με την PROSITE.
DR (Database Reference): Περιέχει όλες τις εγγραφές της SWISS-PROT που ακολουθούν το συγκεκριμένο pattern.
3D (3D Structure): Περιέχει όλες τις εγγραφές της Protein Data Bank που περιέχει τις δομές βιομακρομορίων και ακολουθούν το συγκεκριμένο pattern.
DO (Documentation): Σύνδεσμος για εγγραφή που αναλυτικά στοιχεία σχετικά με τη βιολογική λειτουργία των ακολουθιών που περιέχουν το συγκεκριμένο pattern καθώς και βιβλιογραφικές αναφορές.
//: Δηλώνει το τέλος της εγγραφής.
5. Εγγραφή της PDB για την δομή στο χώρο της Διϋδροφολικής Αναγωγάσης (DHFR) από τον οργανισμό Lactobacillus casei.
HEADER OXIDOREDUCTASE 04-NOV-97 1HFR TITLE COMPARISON OF TERNARY CRYSTAL COMPLEXES OF HUMAN TITLE 2 DIHYDROFOLATE REDUCTASE WITH NADPH AND A CLASSICAL TITLE 3 ANTITUMOR FUROPYRIMDINE COMPND MOL_ID: 1; COMPND 2 MOLECULE: DIHYDROFOLATE REDUCTASE; COMPND 3 CHAIN: NULL; COMPND 4 SYNONYM: DHFR; COMPND 5 EC: 1.5.1.3; COMPND 6 ENGINEERED: YES; COMPND 7 OTHER_DETAILS: COMPLEXED WITH NADPH AND COMPND 8 FURO[2,3D]FUROPYRIMIDINE SOURCE MOL_ID: 1; SOURCE 2 ORGANISM_SCIENTIFIC: HOMO SAPIENS; SOURCE 3 ORGANISM_COMMON: HUMAN; SOURCE 4 EXPRESSION_SYSTEM: ESCHERICHIA COLI; SOURCE 5 EXPRESSION_SYSTEM_STRAIN: JM107 KEYWDS OXIDOREDUCTASE, ONE-CARBON METABOLISM EXPDTA X-RAY DIFFRACTION AUTHOR V.CODY,N.GALITSKY,J.R.LUFT,W.PANGBORN,R.L.BLAKLEY,A.GANGJEE REVDAT 1 28-JAN-98 1HFR 0 JRNL AUTH V.CODY,N.GALITSKY,J.R.LUFT,W.PANGBORN,R.L.BLAKLEY, JRNL AUTH 2 A.GANGJEE JRNL TITL COMPARISON OF TERNARY CRYSTAL COMPLEXES OF HUMAN JRNL TITL 2 DIHYDROFOLATE REDUCTASE WITH NADPH AND A CLASSICAL JRNL TITL 3 ANTITUMOR FUROPYRIMDINE JRNL REF TO BE PUBLISHED JRNL REFN 0353 REMARK 1 REMARK 1 REFERENCE 1 REMARK 1 AUTH V.CODY,N.GALITSKY,J.R.LUFT,W.PANGBORN,A.GANGJEE, REMARK 1 AUTH 2 R.DEVRAJ,S.F.QUEENER,R.L.BLAKLEY REMARK 1 TITL COMPARISON OF TERNARY COMPLEXES OF PNEUMOCYSTIS REMARK 1 TITL 2 CARINII AND WILD-TYPE HUMAN DIHYDROFOLATE REDUCTASE REMARK 1 TITL 3 WITH A NOVEL CLASSICAL ANTITUMOR REMARK 1 TITL 4 FURO[2,3-D]PYRIMIDINE ANTIFOLATE REMARK 1 REF ACTA CRYSTALLOGR.,SECT.D V. 53 638 1997 REMARK 1 REFN ASTM ABCRE6 DK ISSN 0907-4449 0766 . . . REMARK 1 REFERENCE 4 REMARK 1 AUTH V.CODY,J.R.LUFT,E.CISZAK,T.I.KALMAN,J.H.FREISHEIM REMARK 1 TITL CRYSTAL STRUCTURE DETERMINATION AT 2.3 A OF REMARK 1 TITL 2 RECOMBINANT HUMAN DIHYDROFOLATE REDUCTASE TERNARY REMARK 1 TITL 3 COMPLEX WITH NADPH AND METHOTREXATE-GAMMA-TETRAZOLE REMARK 1 REF ANTI-CANCER DRUG DES. V. 7 483 1992 REMARK 1 REFN ASTM ACDDEA UK ISSN 0266-9536 0807 REMARK 2 REMARK 2 RESOLUTION. 2.1 ANGSTROMS. REMARK 3 REMARK 3 REFINEMENT. REMARK 3 PROGRAM : PROFFT REMARK 3 AUTHORS : KONNERT,HENDRICKSON,FINZEL REMARK 3 REMARK 3 DATA USED IN REFINEMENT. REMARK 3 RESOLUTION RANGE HIGH (ANGSTROMS) : NULL REMARK 3 RESOLUTION RANGE LOW (ANGSTROMS) : NULL REMARK 3 DATA CUTOFF (SIGMA(F)) : NULL REMARK 3 COMPLETENESS FOR RANGE (%) : NULL REMARK 3 NUMBER OF REFLECTIONS : NULL REMARK 3 REMARK 3 FIT TO DATA USED IN REFINEMENT. REMARK 3 CROSS-VALIDATION METHOD : NULL REMARK 3 FREE R VALUE TEST SET SELECTION : NULL REMARK 3 R VALUE (WORKING + TEST SET) : NULL REMARK 3 R VALUE (WORKING SET) : 0.1995 . . . REMARK 200 COMPLETENESS FOR RANGE (%) : 84.7 REMARK 200 DATA REDUNDANCY : 3.09 REMARK 200 R MERGE (I) : 0.086 REMARK 200 R SYM (I) : NULL REMARK 200 FOR THE DATA SET : NULL REMARK 200 REMARK 200 IN THE HIGHEST RESOLUTION SHELL. REMARK 200 HIGHEST RESOLUTION SHELL, RANGE HIGH (A) : NULL REMARK 200 HIGHEST RESOLUTION SHELL, RANGE LOW (A) : NULL REMARK 200 COMPLETENESS FOR SHELL (%) : NULL REMARK 200 DATA REDUNDANCY IN SHELL : NULL REMARK 200 R MERGE FOR SHELL (I) : NULL REMARK 200 R SYM FOR SHELL (I) : NULL REMARK 200 FOR SHELL : NULL REMARK 200 REMARK 200 METHOD USED TO DETERMINE THE STRUCTURE: NULL REMARK 200 SOFTWARE USED: NULL REMARK 200 STARTING MODEL: NULL REMARK 200 REMARK 200 REMARK: NULL REMARK 280 REMARK 280 CRYSTAL REMARK 280 SOLVENT CONTENT, VS (%): 48. REMARK 280 MATTHEWS COEFFICIENT, VM (ANGSTROMS**3/DA): 2.54 REMARK 280 REMARK 280 CRYSTALLIZATION CONDITIONS: NULL REMARK 290 REMARK 290 CRYSTALLOGRAPHIC SYMMETRY REMARK 290 SYMMETRY OPERATORS FOR SPACE GROUP: H 3 REMARK 290 REMARK 290 SYMOP SYMMETRY REMARK 290 NNNMMM OPERATOR REMARK 290 1555 X,Y,Z REMARK 290 2555 -Y,X-Y,Z REMARK 290 3555 Y-X,-X,Z REMARK 290 4555 X+2/3,Y+1/3,Z+1/3 REMARK 290 5555 -Y+2/3,X-Y+1/3,Z+1/3 REMARK 290 6555 Y-X+2/3,-X+1/3,Z+1/3 REMARK 290 7555 X+1/3,Y+2/3,Z+2/3 REMARK 290 8555 -Y+1/3,X-Y+2/3,Z+2/3 REMARK 290 9555 Y-X+1/3,-X+2/3,Z+2/3 REMARK 290 REMARK 290 WHERE NNN -> OPERATOR NUMBER REMARK 290 MMM -> TRANSLATION VECTOR REMARK 290 REMARK 290 CRYSTALLOGRAPHIC SYMMETRY TRANSFORMATIONS REMARK 290 THE FOLLOWING TRANSFORMATIONS OPERATE ON THE ATOM/HETATM REMARK 290 RECORDS IN THIS ENTRY TO PRODUCE CRYSTALLOGRAPHICALLY REMARK 290 RELATED MOLECULES. REMARK 290 SMTRY1 1 1.000000 0.000000 0.000000 0.00000 REMARK 290 SMTRY2 1 0.000000 1.000000 0.000000 0.00000 REMARK 290 SMTRY3 1 0.000000 0.000000 1.000000 0.00000 REMARK 290 SMTRY1 2 -0.500000 -0.866082 0.000000 0.00000 REMARK 290 SMTRY2 2 0.865969 -0.500000 0.000000 0.00000 REMARK 290 SMTRY3 2 0.000000 0.000000 1.000000 0.00000 REMARK 290 SMTRY1 3 -0.500000 0.866082 0.000000 0.00000 REMARK 290 SMTRY2 3 -0.865969 -0.500000 0.000000 0.00000 REMARK 290 SMTRY3 3 0.000000 0.000000 1.000000 0.00000 REMARK 290 SMTRY1 4 1.000000 0.000000 0.000000 43.45181 REMARK 290 SMTRY2 4 0.000000 1.000000 0.000000 25.08529 REMARK 290 SMTRY3 4 0.000000 0.000000 1.000000 25.69637 REMARK 290 SMTRY1 5 -0.500000 -0.866082 0.000000 43.45181 REMARK 290 SMTRY2 5 0.865969 -0.500000 0.000000 25.08529 REMARK 290 SMTRY3 5 0.000000 0.000000 1.000000 25.69637 REMARK 290 SMTRY1 6 -0.500000 0.866082 0.000000 43.45181 REMARK 290 SMTRY2 6 -0.865969 -0.500000 0.000000 25.08529 REMARK 290 SMTRY3 6 0.000000 0.000000 1.000000 25.69637 REMARK 290 SMTRY1 7 1.000000 0.000000 0.000000 0.00000 REMARK 290 SMTRY2 7 0.000000 1.000000 0.000000 50.17058 REMARK 290 SMTRY3 7 0.000000 0.000000 1.000000 51.39274 REMARK 290 SMTRY1 8 -0.500000 -0.866082 0.000000 0.00000 REMARK 290 SMTRY2 8 0.865969 -0.500000 0.000000 50.17058 REMARK 290 SMTRY3 8 0.000000 0.000000 1.000000 51.39274 REMARK 290 SMTRY1 9 -0.500000 0.866082 0.000000 0.00000 REMARK 290 SMTRY2 9 -0.865969 -0.500000 0.000000 50.17058 REMARK 290 SMTRY3 9 0.000000 0.000000 1.000000 51.39274 REMARK 290 REMARK 290 REMARK: NULL DBREF 1HFR 1 186 SWS P00374 DYR_HUMAN 1 186 SEQRES 1 186 VAL GLY SER LEU ASN CYS ILE VAL ALA VAL SER GLN ASN SEQRES 2 186 MET GLY ILE GLY LYS ASN GLY ASP LEU PRO TRP PRO PRO SEQRES 3 186 LEU ARG ASN GLU PHE ARG TYR PHE GLN ARG MET THR THR SEQRES 4 186 THR SER SER VAL GLU GLY LYS GLN ASN LEU VAL ILE MET SEQRES 5 186 GLY LYS LYS THR TRP PHE SER ILE PRO GLU LYS ASN ARG SEQRES 6 186 PRO LEU LYS GLY ARG ILE ASN LEU VAL LEU SER ARG GLU SEQRES 7 186 LEU LYS GLU PRO PRO GLN GLY ALA HIS PHE LEU SER ARG SEQRES 8 186 SER LEU ASP ASP ALA LEU LYS LEU THR GLU GLN PRO GLU SEQRES 9 186 LEU ALA ASN LYS VAL ASP MET VAL TRP ILE VAL GLY GLY SEQRES 10 186 SER SER VAL TYR LYS GLU ALA MET ASN HIS PRO GLY HIS SEQRES 11 186 LEU LYS LEU PHE VAL THR ARG ILE MET GLN ASP PHE GLU SEQRES 12 186 SER ASP THR PHE PHE PRO GLU ILE ASP LEU GLU LYS TYR SEQRES 13 186 LYS LEU LEU PRO GLU TYR PRO GLY VAL LEU SER ASP VAL SEQRES 14 186 GLN GLU GLU LYS GLY ILE LYS TYR LYS PHE GLU VAL TYR SEQRES 15 186 GLU LYS ASN ASP HET NAP 187 48 HET MOT 187 32 HETNAM NAP NADP NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE HETNAM MOT N-[4-[(2,4-DIAMINOFURO[2,3D]PYRIMIDIN-5-YL)METHYL] HETNAM 2 MOT METHYLAMINO]-BENZOYL]-L-GLUTAMATE HETSYN NAP 2'-MONOPHOSPHOADENOSINE 5'-DIPHOSPHORIBOSE FORMUL 2 NAP C21 H28 N7 O17 P3 FORMUL 3 MOT C20 H22 N6 O6 FORMUL 4 HOH *35(H2 O1) HELIX 1 1 ARG 28 THR 39 1 12 HELIX 2 2 LYS 54 SER 59 1 6 HELIX 3 3 GLU 62 ASN 64 5 3 HELIX 4 4 LEU 93 GLU 101 1 9 HELIX 5 5 PRO 103 ALA 106 1 4 HELIX 6 6 SER 118 ALA 124 1 7 SHEET 1 A 8 GLN 170 GLU 172 0 SHEET 2 A 8 ILE 175 ASN 185 -1 N TYR 177 O GLN 170 SHEET 3 A 8 HIS 130 ILE 138 -1 N ARG 137 O LYS 178 SHEET 4 A 8 LEU 4 VAL 10 1 N CYS 6 O LYS 132 SHEET 5 A 8 VAL 112 ILE 114 1 N VAL 112 O ASN 5 SHEET 6 A 8 LEU 49 GLY 53 1 N LEU 49 O TRP 113 SHEET 7 A 8 ILE 71 LEU 75 1 N ILE 71 O VAL 50 SHEET 8 A 8 PHE 88 SER 90 1 N PHE 88 O VAL 74 LINK AN6 NAP 187 OE1 GLU 123 CISPEP 1 ARG 65 PRO 66 0 -5.73 CISPEP 2 GLY 116 GLY 117 0 1.43 CRYST1 86.900 86.900 77.090 90.00 90.00 120.00 H 3 9 ORIGX1 1.000000 0.000000 0.000000 0.00000 ORIGX2 0.000000 1.000000 0.000000 0.00000 ORIGX3 0.000000 0.000000 1.000000 0.00000 SCALE1 0.011507 0.006644 0.000000 0.00000 SCALE2 0.000000 0.013288 0.000000 0.00000 SCALE3 0.000000 0.000000 0.012972 0.00000 ATOM 1 N VAL 1 9.276 20.928 2.652 1.00 36.50 N ATOM 2 CA VAL 1 8.254 21.786 1.963 1.00 36.90 C ATOM 3 C VAL 1 8.075 21.174 0.564 1.00 35.28 C ATOM 4 O VAL 1 6.989 20.612 0.266 1.00 35.71 O ATOM 5 CB VAL 1 8.615 23.228 2.320 1.00 38.57 C ATOM 6 CG1 VAL 1 9.294 24.255 1.444 1.00 38.43 C ATOM 7 CG2 VAL 1 7.447 24.037 2.968 1.00 38.75 C ATOM 8 N GLY 2 9.056 21.228 -0.264 1.00 33.51 N ATOM 9 CA GLY 2 9.098 20.717 -1.627 1.00 32.93 C ATOM 10 C GLY 2 9.472 19.238 -1.640 1.00 30.93 C ATOM 11 O GLY 2 9.017 18.424 -0.829 1.00 31.87 O . . . . . ATOM 1494 N ASP 186 4.926 5.677 -4.032 1.00 38.26 N ATOM 1495 CA ASP 186 4.210 4.604 -3.371 1.00 39.36 C ATOM 1496 C ASP 186 3.878 4.874 -1.906 1.00 39.93 C ATOM 1497 O ASP 186 3.546 3.740 -1.459 1.00 41.80 O ATOM 1498 CB ASP 186 5.110 3.312 -3.390 1.00 39.19 C ATOM 1499 CG ASP 186 6.158 3.373 -2.283 1.00 39.17 C ATOM 1500 OD1 ASP 186 6.189 4.348 -1.493 1.00 38.79 O ATOM 1501 OD2 ASP 186 6.971 2.424 -2.156 1.00 39.06 O ATOM 1502 OXT ASP 186 3.944 5.902 -1.229 1.00 40.01 O TER 1503 ASP 186 HETATM 1504 AP NAP 187 26.498 8.553 8.777 1.00 20.52 P HETATM 1505 AO1 NAP 187 27.005 9.743 8.234 1.00 21.63 O HETATM 1506 AO2 NAP 187 25.178 8.109 8.289 1.00 22.30 O HETATM 1507 AO5* NAP 187 26.717 8.412 10.391 1.00 24.11 O HETATM 1508 AC5* NAP 187 25.987 7.510 11.292 1.00 25.77 C HETATM 1509 AC4* NAP 187 25.711 8.495 12.492 1.00 27.24 C HETATM 1510 AO4* NAP 187 24.773 9.267 12.190 1.00 28.36 O HETATM 1511 AC3* NAP 187 24.927 7.457 13.485 1.00 26.85 C HETATM 1512 AO3* NAP 187 26.125 6.976 14.298 1.00 29.04 O HETATM 1513 AC2* NAP 187 24.090 8.492 14.215 1.00 28.80 C HETATM 1514 AO2* NAP 187 24.877 9.337 14.969 1.00 26.09 O HETATM 1515 AC1* NAP 187 23.593 9.346 12.969 1.00 28.23 C HETATM 1516 AN9 NAP 187 22.404 8.860 12.331 1.00 30.46 N HETATM 1517 AC8 NAP 187 22.437 8.209 11.136 1.00 31.29 C HETATM 1518 AN7 NAP 187 21.274 7.825 10.729 1.00 31.20 N . . . HETATM 1545 NC4 NAP 187 27.789 10.072 0.891 1.00 21.30 C HETATM 1546 NC5 NAP 187 27.922 10.049 2.381 1.00 20.60 C HETATM 1547 NC6 NAP 187 29.052 9.696 2.993 1.00 22.66 C HETATM 1548 AP2* NAP 187 25.004 8.883 16.659 1.00 25.53 P HETATM 1549 AOP1 NAP 187 23.598 8.742 17.048 1.00 24.21 O HETATM 1550 AOP2 NAP 187 25.661 10.186 17.021 1.00 25.77 O HETATM 1551 AOP3 NAP 187 25.896 7.761 16.613 1.00 25.55 O HETATM 1552 N1 MOT 187 28.839 13.061 -4.527 1.00 18.22 N HETATM 1553 C2 MOT 187 27.545 12.810 -4.561 1.00 17.16 C . . . . HETATM 1581 CD MOT 187 33.609 21.950 -4.346 1.00 35.15 C HETATM 1582 OE1 MOT 187 33.967 20.785 -4.314 1.00 36.23 O HETATM 1583 OE2 MOT 187 34.133 22.820 -4.999 1.00 37.19 O HETATM 1584 O HOH 188 32.950 12.394 -5.974 1.00 32.65 O HETATM 1585 O HOH 189 24.499 11.873 -8.062 1.00 20.17 O HETATM 1586 O HOH 190 22.883 12.007 -10.563 1.00 21.65 O . . . HETATM 1617 O HOH 221 39.580 2.776 -1.691 1.00 15.00 O HETATM 1618 O HOH 222 18.655 21.208 -7.395 1.00 15.00 O CONECT 970 969 1521 CONECT 1504 1505 1506 1507 1526 CONECT 1505 1504 CONECT 1506 1504 CONECT 1507 1504 1508 . . CONECT 1580 1579 1581 CONECT 1581 1580 1582 1583 CONECT 1582 1581 CONECT 1583 1581 MASTER 228 0 2 6 8 0 0 6 1617 1 81 15 END
Επεξήγηση πεδίων μιας εγγραφής PDB
HEADER: Περιέχει ένα τετραψήφιο κωδικό για την αναγνώριση της εγγραφής στην PDB, μια γενική ταξινόμηση του μακρομορίου καθώς και την ημερομηνία κατάθεσης της δομής στην Protein Data Bank.
TITLE: Τίτλος που περιλαμβάνει συνήθως τα περιεχόμενα της εγγραφής, τι είδους πειραματική διαδικασία χρησιμοποιήθηκε, ύπαρξη μεταλλάξεων. Επιτρέπει στον ερεύνητη που κατέθεσε τη δομή να καταδείξει τη σημαντικότητα της εργασίας αυτής.
COMPOUND: Το πεδίο compound περιέχει πληροφορίες για το μακρομόριο που αναφέρεται στη δομή καθώς και τα άλλα μόρια (μικρές οργανικές ενώσεις, μέταλλα) με τα οποία έχει τυχόν συμπλοκοποιηθεί.
SOURCE: Βιολογική προέλευση του μακρομορίου που αναφέρεται στην εγγραφή.
KEYWDS: Χαρακτηριστικές λέξεις-κλειδιά για τον χαρακτηρισμό της εγγραφής.
EXPDTA: Πειραματική τεχνική για τον προσδιορισμό της δομής (X-Ray Crystallography/NMR/Theoretical Model).
AUTHOR: Λίστα με τα ονόματα των ερευνητών που συμμετείχαν στον προσδιορισμό της δομής.
JRNL: Πρωταρχική βιβλιογραφική αναφορά η οποία αναφερέται στον προσδιορισμό της δομής που αναφέρεται στην συγκεκριμένη εγγραφή.
REMARK: Το πεδίο REMARK περιλαμβάνει μια σειρά από πληροφορίες σχετικές με την κατατεθειμένη δομή.
Καταρχήν περιέχει βιβλιογραφικές αναφορές που σχετίζονται άμεσα με το προς μελέτη μακρομόριο.
Στο πεδίο REMARK περιλαμβάνονται και στοιχεία σχετικά με την πειραματική διαδικασία που ακολουθήθηκε για την λύση της δομής όπως είναι τα προγράμματα που χρησιμοποιήθηκαν, οι τιμές διαφόρων δεικτών, γενικά πληροφορίες που αποδεικνύουν την ορθότητα της δομής.
SEQRES: Περιέχει την αλληλουχία του προς μελέτη μακρομορίου. Για τις πρωτεΐνες ακολουθείται ο κώδικας των 3 γραμμάτων.
HET: Αναφέρεται στα μόρια (ετεροάτομα) που δεν είναι αμινοξέα ή νουκλεοτίδια. Αυτά μπορεί να είναι προσθετικές ομάδες και ιόντα για τα οποία έχουν προσδιοριστεί οι συντεταγμένες τους. Τα στοιχεία που δίνονται για αυτά είναι ένας κωδικός για να διευκρινίζονται σε σχέση με τα άλλα κατάλοιπα της εγγραφής, η αρίθμηση που έχουν μέσα στο αρχείο των συντεταγμένων και τέλος ο αριθμός των ατόμων από τα οποία αποτελούνται.
ΗΕΤΝΑΜ: Ονοματολογία των καταλοίπων που περιέχονται στο πεδίο HET.
FORMUL: Μοριακός τύπος των καταλοίπων που αναφέρονται στο πεδίο HET.
HELIX: Τμήματα της ακολουθίας που έχουν ελικοειδή δομή.
SHEET: Τμήματα της ακολουθίας που έχουν εκτεταμένη δομή.
CRYST1: Περιέχει τις παραμέτρους μοναδιαίας κυψελλίδας και την ομάδα συμμετρίας χώρου.
ORIGXn(n=1..3): Πίνακας Μετατροπής από σύστημα ορθογωνίων συντεταγμένων στις συντεταγμένες που κατατέθηκαν αρχικά στην PDB.
SCALEn: Πίνακας Μετατροπής από σύστημα ορθογωνίων συντεταγμένων στις κρυσταλλογραφικές συντεταγμένες.
ATOM: Περιέχει τις συντεταγμένες των ατόμων στους άξονες Χ, Υ, Ζ. Περιλαμβάνει επίσης και άλλα στοιχεία όπως τα άτομα για τα οποία αναφέρονται οι συντεταγμένες και σε ποια κατάλοιπα ανήκουν. Πρέπει να σημειωθεί ότι κάθε είδους δεδομένο που περιέχεται στο πεδίο ΑΤΟΜ είναι τοποθετημένο σε καθορισμένες θέσεις (στήλες) της εγγραφής όπως αυτές παρουσιάζονται παρακάτω:
ΣΤΗΛΕΣ Περιεχόμενα κάθε στήλης --------------------------------------------------------------------------------- 1 - 6 "ATOM " δηλώνει ότι πρόκειται για το πεδίο ΑΤΟΜ. 7 - 11 Αύξων αριθμός του ατόμου. 13 - 16 Τύπος ατόμου. 18 - 20 Όνομα καταλοίπου. Για τα αμινοξέα ακολουθείται ο κώδικας των 3 γραμμάτων. 22 (chainΙD) Χαρακτήρας που ταυτοποιεί την αλυσίδα, αν περιέχονται περισσότερες από μια στην εγγραφή. 23 - 26 Αρίθμηση του καταλοίπου στην αλυσίδα 31 - 38 x Συντεταγμένες ατόμου (σε Angstroms) στον άξονα Χ σε τρισορθογώνιο σύστημα αξόνων. 39 - 46 y Συντεταγμένες ατόμου (σε Angstroms) στον άξονα Y σε τρισορθογώνιο σύστημα αξόνων. 47 - 54 z Συντεταγμένες ατόμου (σε Angstroms) στον άξονα Z σε τρισορθογώνιο σύστημα αξόνων. 55 - 60 Συντελεστής κατάληψης(occupancy) 61 - 66 Παράγοντας θερμοκρασίας(Temperature factor) 77 - 78 Σύμβολο του ατόμου. 79 - 80 Φορτίο του ατόμου (Αν υπάρχει).
TER: Το πεδίο ΤΕR δηλώνει το τέλος της παράθεσης των ατόμων που απαρτίζουν μια αλυσίδα.
HETATM: Συντεταγμένες των ετεροατόμων. Η μορφοποίηση παρουσίασης τους ακολουθεί του ίδιους κανόνες με το πεδίο ΑΤΟΜ.
CONECT: Το πεδίο CONECT καθορίζει τα άτομα τα οποία συμμετέχουν στον σχηματισμό δεσμών. Κάθε άτομο συμβολίζεται με την αρίθμηση του όπως είναι καθορισμένη στα πεδία ΑΤΟΜ.
MASTER: Αποτελεί ένα πεδίο που χρησιμοποιείται για μια απλή οργάνωση της εγγραφής. Πρόκειται για μια σειρά από αριθμούς που δεν είναι τίποτε άλλο από το άθροισμα των γραμμών για συγκεκριμένα πεδία της εγγραφής.
END: Υποδηλώνει τη λήξη της εγγραφής.