ΒΙΟΦΥΣΙΚΗ
ΑΣΚΗΣΗ:
ΜΕΡΟΣ Α:ΒΑΣΕΙΣ ΔΕΔΟΜΕΝΩΝ ΔΟΜΙΚΗΣ ΒΙΟΛΟΓΙΑΣ ΚΑΙ ΕΡΓΑΛΕΙΑ ΑΝΑΛΥΣΗΣ ΚΑΙ ΠΡΟΒΛΕΨΗΣ
ΠΡΩΤΕΪΝΙΚΩΝ ΔΟΜΩΝ
ΜΕΡΟΣ Β:ΧΑΡΑΚΤΗΡΙΣΜΟΣ, ΚΑΤΗΓΟΡΙΟΠΟΙΗΣΗ ΚΑΙ ΠΡΟΓΝΩΣΗ ΔΟΜΗΣ ΠΡΩΤΕΪΝΩΝ
Μ. Καρμιράντζου, Β. Ι. Προμπονάς
και
Σ. Ι. Χαμόδρακας
Τμήμα Βιολογίας,
Τομέας Βιολογίας Κυττάρου και Βιοφυσικής,
Πανεπιστήμιο Αθηνών
Αθήνα, Απρίλιος 2000
ΕΙΣΑΓΩΓΗ
ΓΕΝΙΚΑ
Μιλήσαμε ήδη για βάσεις βιολογικών δεδομένων παρόλα αυτά το κεφάλαιο αυτό δεν θα ήταν ολοκληρωμένο αν δεν κάναμε κάποια αναφορά στα συστήματα κατηγοριοποίησης των πρωτεϊνικών δομών (Protein Structure Classification Schemes).
Παρόλο που ο αριθμός των πρωτεϊνικών δομών, που εναποτίθενται στην βάση δεδομένων Protein Data Bank (PDB) από μεθόδους κρυσταλλογραφίας ακτίνων-Χ σε ατομική ή περίπου ατομική διακριτικότητα και φασματοσκοπία NMR, είναι σχετικά μικρός (~10.000), οι πληροφορίες που εμπεριέχουν γίνονται όλο και πιο σημαντικές με την αποκάλυψη και νέων συνδυασμών δευτεροταγούς δομής. Περίπου 200 νέες πρωτεΐνες κατατίθενται μηνιαίως στην PDB.
Πολλές πρωτεΐνες μοιράζονται δομικά στοιχεία και ομοιότητες που αντανακλούν ίσως, σε κάποιες περιπτώσεις, την εξελικτική τους προέλευση. Η ίδια η διαδικασία της εξέλιξης περιλαμβάνει αντικαταστάσεις, εισαγωγές ή / και αφαιρέσεις αμινοξέων από την ακολουθία των βιολογικών μακρομορίων. Στις πρωτεΐνες που έχουν μακρινή εξελικτική σχέση, οι αλλαγές αυτές μπορεί να είναι εκτενείς με αποτέλεσμα να διπλώνουν κατά τέτοιο τρόπο που και ο αριθμός αλλά και ο προσανατολισμός των στοιχείων δευτεροταγούς δομής να ποικίλει σε μεγάλο βαθμό. Παρόλα αυτά, όταν για παράδειγμα, οι λειτουργίες ορισμένων πρωτεϊνών έχουν συντηρηθεί, τα δομικά περιβάλλοντα των αμινοξέων των ενεργών κέντρων είναι επίσης διατηρημένα. Βλέπουμε λοιπόν ότι για να κατανοήσουμε το σύνολο όλων αυτών των τόσων διαφορετικών μα και τόσων όμοιων, σε μερικές περιπτώσεις δομών, είναι ανάγκη να αντιπαραβάλλουμε, σχολιάσουμε αλλά και να κατηγοριοποιήσουμε όλες τις προσδιορισμένες πρωτεΐνες. Έτσι ίσως ερμηνεύσουμε καλύτερα τις σχέσεις μεταξύ αλληλουχίας, δομής και λειτουργίας αλλά και τις βαθύτερες εξελικτικές / φυλογενετικές διαδικασίες που έχουν οδηγήσει στην δημιουργία αυτών των ποικιλόμορφων δομικών σχηματισμών.
Γίνεται εύκολα αντιληπτό ότι το είδος πληροφοριών που παρουσιάζονται σ'ένα σύστημα ταξινόμησης πρωτεϊνικών δομών είναι αποκλειστικά εξαρτώμενο από την φιλοσοφία της μεθόδου και άρα και από τις μεθόδους που χρησιμοποιήθηκαν για την αναγνώριση και εκτίμηση των διαφορών και των ομοιοτήτων μεταξύ διαφόρων δομών.
ΔΟΜΙΚΑ ΣΤΟΙΧΕΙΑ ΠΡΩΤΕΪΝΩΝ ΚΑΙ ΣΥΣΤΗΜΑΤΑ ΚΑΤΗΓΟΡΙΟΠΟΙΗΣΗΣ ΤΩΝ ΤΡΙΣΔΙΑΣΤΑΤΩΝ ΔΟΜΩΝ ΤΟΥΣ
Από το 1959 που προσδιορίσθηκε η πρώτη τρισδιάστατη δομή της μυοσφαιρίνης μέχρι τώρα, η πολυπλοκότητα και η ποικιλομορφία των πρωτεϊνικών δομών αυξάνεται παράλληλα με τον αριθμό των νέων προσδιοριζόμενων μακρομορίων. Έτσι, από τότε μέχρι σήμερα, έχει αναγνωρισθεί ότι οι πρωτεΐνες αποτελούνται από αυτοτελή ημιανεξάρτητα δομικά στοιχεία που μπορούν να είναι συνεχή ή μη συνεχή στην πρωτεϊνική ακολουθία (Domains). Έτσι, προφανώς, το πρώτο βήμα για την κατάταξη των πρωτεϊνών σε τάξεις, κλάσεις, οικογένειες κλπ είναι η ταυτοποίηση των δομικών στοιχείων τους.
Δυο από τα πιο διαδεδομένα συστήματα κατηγοριοποίησης πρωτεϊνικών δομών παρουσιάζονται παρακάτω.
SCOP
Η βάση πρωτεϊνικών δομών SCOP (Structural Classification of Proteins) που διατηρείται στο κέντρο ερευνών Μοριακής Βιολογίας και Πρωτεϊνικής Μηχανικής στο Cambridge της Αγγλίας (MRC), περιγράφει τις δομικές και εξελικτικές σχέσεις μεταξύ πρωτεϊνών με γνωστή δομή (Murzin et al, 1995). Επειδή ακόμα και τα πιο σύγχρονα «αυτόματα εργαλεία» για την σύγκριση των στοιχείων δευτεροταγούς δομής δεν μπορούν αξιόπιστα να αναγνωρίσουν όλες τις σχέσεις μεταξύ των πρωτεϊνικών δομών, το SCOP κατασκευάζεται χρησιμοποιώντας άμεσα εποπτικά κριτήρια και την εμπειρία ειδικών αναλυτών. Το εγχείρημα αυτό είναι περίπλοκο μιας και οι πρωτεϊνικές δομές επιδεικνύουν μια τεράστια ποικιλία σχηματισμών που κυμαίνεται από μικρά μοναδιαία δομικά στοιχεία ως και τεράστια σύμπλοκα πολλαπλών domains. Σε μερικές μάλιστα περιπτώσεις, έχει σημασία να αναλύουμε μια πρωτεϊνική δομή στο σύνολο της αλλά και στο επίπεδο των μεμονωμένων αυτοτελών δομικών στοιχείων της.
SCOP classification
Οι πρωτείνες ταξινομούνται κατά ένα ιεραρχικό τρόπο ο οποίος αντανακλά την δομική και την εξελικτική τους σχέση. Υπάρχουν πολλά και διαφορετικά επίπεδα στην ιεραρχία, αλλά κατά κύριο λόγο αυτά περιγράφουν τις έννοιες «Οικογένεια» (Family), «Υπέρ-οικογένεια» (Superfamily) και «Δίπλωμα» (Fold). Τα όρια μεταξύ των επιπέδων αυτών μπορεί να είναι υποκειμενικά αλλά κατά έναν γενικό τρόπο τα ανώτερα επίπεδα αντανακλούν και πιο κοντινές δομικές ομοιότητες. Τα πρώτα δυο επίπεδα περιγράφουν κοντινές ή/και μακρινές εξελικτικές σχέσεις ενώ στο τρίτο επίπεδο αναπαριστώνται οι γεωμετρικές ομοιότητες των στοιχείων δευτεροταγούς δομής των πρωτεϊνών.
Πιο συγκεκριμένα, η κατηγοριοποίηση των πρωτεϊνών με βάση το σύστημα SCOP γίνεται με βάση τα παρακάτω κριτήρια:
«Οικογένεια» (Family). Οι πρωτεΐνες ομαδοποιούνται στην ίδια οικογένεια με βάση τις εξελικτικές τους συγγένειες; αν δηλαδή οι αμινοξικές ακολουθίες των πρωτεϊνών είναι όμοιες μεταξύ τους σε ποσοστό μεγαλύτερο από 30% (Sequence Identity >= 30%) τότε αποτελούν μέλη της ίδιας οικογένειας.
«Υπερ-οικογένεια» (Superfamily). Οι πρωτεΐνες ταξινομούνται στην ίδια υπερ-οικογένεια άν με βάση τα δομικά και λειτουργικά χαρακτηριστικά τους μπορούμε να τους αποδώσουμε κάποια κοινή εξελικτική σχέση.
«Δίπλωμα» (Fold). Πρωτεΐνες οι οποίες προέρχονται από διαφορετικές οικογένειες ή/και υπερ-οικογένειες ανήκουν σε μια κοινή ομάδα (διπλώματος) εάν έχουν τα ίδια στοιχεία δευτεροταγούς δομής στην ίδια οργάνωση και με την ίδια τοπολογία.
«Τάξη» (Class). Τέσσερις κύριες δομικές κατηγορίες πρωτεϊνών έχουν ταυτοποιηθεί με βάση τον τρόπο που διπλώνουν τα στοιχεία δευτεροταγούς δομής τους:
a) all-α, η δομή ουσιαστικά σχηματίζεται από α-έλικες;
b) all-β, η δομή αποτελείται από β-πτυχωτές επιφάνειες;
γ) α/β, α-έλικες και β-πτυχωτές επιφάνειες εναλλάσσονται στην δομή της πρωτεΐνης; και
δ) α+β, α-έλικες και β-πτυχωτές επιφάνειες βρίσκονται σε διακριτές περιοχές της δομής .
Η ιεραρχική διαμόρφωση του SCOP παρουσιάζεται στην εικόνα 1 για την πρωτεϊνική δομή της ανθρώπινης αιμοσφαιρίνης (κωδικός PDB : 2hhb)
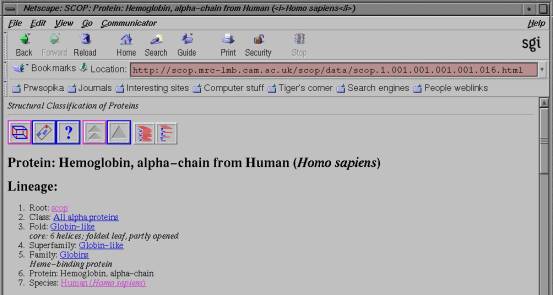
Το σύστημα SCOP είναι ελεύθερα διαθέσιμο για αναζήτηση στην ηλεκτρονική διεύθυνση: http://scop.mrc-lmb.cam.ac.uk/scop/
CATH
Η βάση δεδομένων CATH (Orengo et al., 1997), που διατηρείται στο Πανεπιστήμιο UCL του Λονδίνου, περιέχει μια ιεραρχική ταξινόμηση των αυτοτελών δομικών στοιχείων (domains) των πρωτεϊνικών δομών που είναι κατατεθειμένες στην PDB, και οι οποίες έχουν προσδιοριστεί σε διακριτικότητα καλύτερη από 3 A. Μη πρωτεϊνικές δομές, και Cα δομές δεν έχουν συμπεριληφθεί. Το σύστημα CATH με την εφαρμογή μια σειράς αυτοματοποιημένων μεθόδων παρέχει εξειδικευμένες πληροφορίες για την κατηγοριοποίηση των πρωτεϊνικών domains. Παρόλα αυτά, εμπειρικά ανθρώπινα κριτήρια χρησιμοποιούνται για τις εξειδικευμένες περιπτώσεις που τα «αυτόματα εργαλεία» σύγκρισης των πρωτεϊνικών δομών δεν δίνουν ακριβή ή έγκυρα αποτελέσματα. Κατ' αναλογία με το σύστημα ταξινόμησης των ενζύμων (E.C. system) κάθε κατηγορία πρωτεϊνικών δομών χαρακτηρίζεται από ένα μοναδικό αριθμό και ταυτοποιείται από ένα περιγραφικό όνομα. Το σχήμα αυτό επιτρέπει και κατάλληλους χειρισμούς των πληροφοριών.
Πέντε κύρια επίπεδα χρησιμοποιούνται στην ταξινόμηση αυτή: τα επίπεδα Τάξης (Class), Αρχιτεκτονικής (Architecture), Τοπολογίας (Topology), Ομόλογης Υπέρ-οικογένειας (Homologous superfamily) και Αλληλουχίας (Sequence).
«Τάξη» (Class). Η τάξη προσδιορίζεται από το ποσοστό των στοιχείων δευτεροταγούς δομής μιας πρωτεΐνης και το πακετάρισμα τους. Τέσσερις κύριες ομάδες πρωτεϊνικών δομικών στοιχείων έχουν ταυτοποιηθεί : (i) mainly-α, (ii) mainly-β, (iii) α-β, που περιλαμβάνει εναλλασσόμενες α/β και α+β δομές, και (iv) δομές με χαμηλό ποσοστό δευτεροταγούς δομής.
«Αρχιτεκτονική» (Architecture) περιγράφει την οργάνωση των στοιχείων δευτεροταγούς δομής χωρίς να λαμβάνεται υπόψη ο τρόπος με τον οποίον αυτά συνδέονται (π.χ. βαρέλι (barrel), σάντουιτς (sandwich) κ.α.)
«Τοπολογία» (Topology) δίνει μια περιγραφή του ολικού σχήματος και του τρόπου σύνδεσης των στοιχείων δευτεροταγούς δομής. Με βάση μια σειρά από εμπειρικές παραμέτρους και χρησιμοποιώντας γρήγορους αλγόριθμους που επιτυγχάνουν την ολική σύγκριση των δομικών στοιχείων, οι πρωτεΐνες ομαδοποιούνται σε διαφορετικές κατηγορίες τοπολογίας. Δομές που κατά το 60% μοιάζουν με την μικρότερη ταξινομούνται στο ίδιο επίπεδο τοπολογίας.
«Ομόλογη οικογένεια» (Homology superfamily) ομαδοποιεί όλα εκείνα τα δομικά στοιχεία που έχουν 35% ομοιότητα μεταξύ τους στο επίπεδο της αλληλουχίας τους και συνεπώς πιστεύεται ότι προέρχονται από ένα κοινό πρόγονο, δηλαδή είναι ομόλογες πρωτεΐνες.
«Αλληλουχία» (Sequence) παρέχει το τελευταίο επίπεδο ιεράρχησης όπου οι δομές μιας ομόλογης οικογένειας ταξινομούνται περαιτέρω με βάση την ομοιότητα των αμινοξικών αλληλουχιών τους. Στο επίπεδο αυτό, τα διάφορα domains χαρακτηρίζονται από ποσοστό μεγαλύτερο από 30% ομοιότητας των αλληλουχιών τους (Sequence Identity >= 30%) γεγονός που υποδηλώνει παρόμοιες δομές, άρα και λειτουργίες.
Το CATH είναι ελεύθερα προσβάσιμο για αναζήτηση με λέξεις κλειδιά (είτε όνομα-κατηγορία πρωτεϊνών είτε συγκεκριμένο κωδικό πρωτεΐνης στην βάση δεδομένων πρωτεϊνικών δομών PDB (π.χ. 1ECA)) μέσω του διαδικτυακού φυλλομετρητή (Web browser) που εδρεύει στο UCL (URL: http://www.biochem.ucl.ac.uk/bsm/cath/ ).
Ένα παράδειγμα του συστήματος CATH δίνεται στην παρακάτω εικόνα για την δομή της ερυθροκρουορίνης (κωδικός PDB :1ECA).
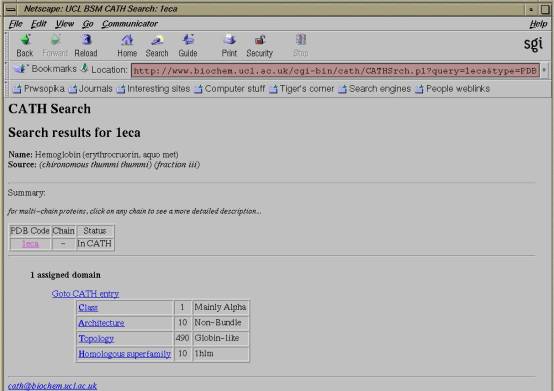
ΠΡΑΚΤΙΚΟ ΜΕΡΟΣ
ΜΕΡΟΣ Α: ΒΑΣΕΙΣ ΔΕΔΟΜΕΝΩΝ ΔΟΜΙΚΗΣ ΚΑΙ ΜΟΡΙΑΚΗΣ ΒΙΟΛΟΓΙΑΣ ΣΤΟ ΔΙΑΔΙΚΤΥΟ (INTERNET)
ΠΡΟΣΒΑΣΗ ΣΤΑ ΔΕΔΟΜΕΝΑ
Στο πρώτο μέρος της άσκησης θα έχουμε πρόσβαση, μέσω του διαδικτύου, σε βάσεις δεδομένων, από τις οποίες θα έχουμε την ευκαιρία να 'αντλήσουμε' κάποιες πρωτεϊνικές ακολουθίες και τις δομές τους. Στη συνέχεια, θα επεξεργαστούμε την πρωτογενή αυτή πληροφορία με προγράμματα λογισμικού (εργαλεία ανάλυσης - analysis tools), που είναι επίσης ελεύθερα προσβάσιμα μέσω του Internet.
Αρχικά, γίνεται η επιλογή της ακολουθίας που θα επεξεργαστεί η κάθε ομάδα. Η άγνωστη ακολουθία κάθε ομάδας βρίσκεται σε FASTA-format στον κατάλογο:
C:\Sequences\[agnosti].seq
όπου '[agnosti.seq]' είναι ακολουθία άγνωστης προέλευσης και λειτουργίας.
Με το φυλλομετρητή δικτύου που διαθέτουμε (Internet Explorer, Netscape, κλπ) επισκεπτόμαστε την κεντρική σελίδα που ετοιμάστηκε για τη διεξαγωγή της άσκησης στη διεύθυνση (URL):
http://biophysics.biol.uoa.gr/doc/links.html
στην οποία είναι δυνατόν να επανερχόμαστε επιλέγοντας 'Home' ή 'Αρχική' σελίδα, ανάλογα με το φυλλομετρητή (Web-Browser).
Συνεχίζουμε τη φυλλομέτρησή μας στα Ελληνικά.
ΑΝΑΖΗΤΗΣΗ ΟΜΟΛΟΓΙΑΣ
Ένα πρώτο βήμα στη μελέτη μιας 'άγνωστης' πρωτεΐνης είναι η αναζήτηση πιθανών ομολογιών μέσα στις βάσεις πρωτεϊνικών δεδομένων.
Από τις επιλογές 'Εργαλεία Ανάλυσης Ακολουθιών' επιλέγουμε το BLAST (Basic Local Alignment Search Tool, Altschul, et al. 1990) και πραγματοποιούμε 'Απλή Αναζήτηση' με το BLAST2.0, έναντι της 'Swissprot', η οποία περιέχει τη μαθηματική ένωση (χωρίς διπλές καταχωρήσεις) των καταχωρήσεων σημαντικών βάσεων πρωτεϊνικών δεδομένων.
ΠΑΡΑΤΗΡΗΣΕΙΣ
> Η ακολουθία πρέπει να εισαχθεί σε FASTA format.
> Είναι σημαντικό να γίνει κατανοητή η διαφορά των αποτελεσμάτων όταν έχει επιλεχθεί 'ungapped alignment'.
> Η προεπιλογή φίλτρου για περιοχές χαμηλής πολυπλοκότητας κάνει περισσότερο ευαίσθητο και αποδοτικό τον αλγόριθμο και καλό είναι να παραμείνει επιλεγμένο.
Παράδειγμα αποτελέσματος για την DYR_LACCA (Query) σε αντιπαραβολή με τον εαυτό της (Subject):
Score = 310 bits (785), Expect = 4e-84
Identities = 148/162 (91%), Positives = 148/162 (91%)
Query: 1 TAFLWAQDRDGLIGKDGHLPWHLPDDLHYFRAQTVGKIMVVGRRTYESFPKRPLPERTNV 60
TAFLWAQDRDGLIGKDGHLPWHLPDDLHYFRAQTVGKIMVVGRRTYESFPKRPLPERTNV
Sbjct: 2 TAFLWAQDRDGLIGKDGHLPWHLPDDLHYFRAQTVGKIMVVGRRTYESFPKRPLPERTNV 61
Query: 61 VLTHQEDYQAQGXXXXXXXXXXXXXXKQHLDQELVIAGGAQIFTAFKDDVDTLLVTRLAG 120
VLTHQEDYQAQG KQHLDQELVIAGGAQIFTAFKDDVDTLLVTRLAG
Sbjct: 62 VLTHQEDYQAQGAVVVHDVAAVFAYAKQHLDQELVIAGGAQIFTAFKDDVDTLLVTRLAG 121
Query: 121 SFEGDTKMIPLNWDDFTKVSSRTVEDTNPALTHTYEVWQKKA 162
SFEGDTKMIPLNWDDFTKVSSRTVEDTNPALTHTYEVWQKKA
Sbjct: 122 SFEGDTKMIPLNWDDFTKVSSRTVEDTNPALTHTYEVWQKKA 163
|
Η τιμή Expect είναι ένα μέτρο αξιοπιστίας και εκφράζει την πιθανότητα η ομοιότητα που παρατηρείται να είναι τυχαία. Τιμές < e-6 θεωρούνται αρκετά αξιόπιστες ενδείξεις πραγματικής ομολογίας. Η τιμή Identities δείχνει το ποσοστό ταυτόσημων καταλοίπων, ενώ η Positives το συνολικό ποσοστό ταυτόσημων ή παρόμοιων καταλοίπων.
Ερώτηση:
Να εντοπίσετε τα αποτελέσματα ομολογίας της άγνωστης ακολουθίας που έχετε ανά ομάδα.
Μπορείτε να αναγνωρίστε την οικογένεια πρωτεϊνών στην οποία ανήκει η άγνωστη ακολουθία σας.
Αναζητήστε τις τέσσερις πιο ομόλογες ακολουθίες (με το υψηλότερο σκορ ομολογίας αλλά και με τιμές Ε-value που να θεωρούνται αρκετά αξιόπιστες ενδείξεις πραγματικής ομολογίας).
Σώστε τα αποτελέσματα της αναζήτησης (τις τέσσερις πιο ομόλογες ακολουθίες σας) σε FASTA format και σε ένα αρχείο με το όνομα 'blast.txt' στον κατάλογο 'C:\sequences\'. και στη συνέχεια επιστρέφετε στην αρχική σελίδα.
ΠΟΛΛΑΠΛΗ ΣΤΟΙΧΙΣΗ ΠΡΩΤΕΪΝΙΚΩΝ ΑΚΟΛΟΥΘΙΩΝ ΚΑΙ ΟΜΟΛΟΓΟΠΟΙΗΣΗ
Με το φυλλομετρητή δικτύου που διαθέτουμε (Internet Explorer, Netscape, κλπ) επισκεπτόμαστε την διεύθυνση (URL):
http://www.ebi.ac.uk/blast2/
στην οποία είναι δυνατόν να χρησιμοποιηθεί η μέθοδος ClustalW για την πολλαπλή στοίχιση ακολουθιών και την αναγνώριση ομόλογων περιοχών τους.
Τώρα μπορούμε να πραγματοποιήσουμε την στοίχιση των ακολουθιών μας. Αρχικά, επιλέγουμε έναν τίτλο για το αποτέλεσμα μας και από τις επιλογές ¨Colour Alignment¨ επιλέγουμε ¨yes¨. Τροφοδοτώντας τις περιοχές κειμένου που μας παρέχονται με τις ακολουθίες των πρωτεϊνών που μας ενδιαφέρουν και ξεκινώντας την εφαρμογή (κουμπί RUN CLUSTALW) ξεκινάμε την εφαρμογή του προγράμματος.
Παράδειγμα για την εφαρμογή της μεθόδου δίνεται παρακάτω για την στοίχιση τριών ακολουθιών που ανήκουν στην οικογένεια των cold-shock πρωτεϊνών.
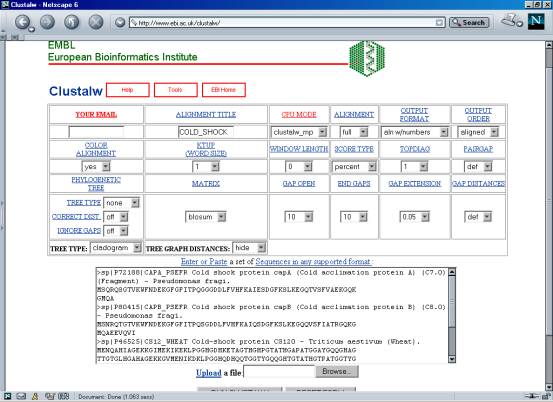
ΠΑΡΑΤΗΡΗΣΕΙΣ
> Οι ακολουθίες πρέπει να εισαχθούν σε FASTA format.
> Συντηρημένα αμινοξέα σημειώνονται με ¨*¨ ενώ κατάλοιπα που είναι μερικώς διατηρημένα μεταξύ των ακολουθιών υπογραμμίζονται με ¨-¨.
Ερώτηση:
Εντοπίσετε ή όχι ομόλογες περιοχές μεταξύ των ακολουθιών σας ; Αν ναι, σε ποια σημεία επί της ακολουθίας βρίσκονται οι περιοχές αυτές ; Αν όχι, μπορείτε να δώσετε μια πιθανή εξήγηση ;
ΣΥΣΤΗΜΑΤΑ ΚΑΤΗΓΟΡΙΟΠΟΙΗΣΗΣ ΠΡΩΤΕΪΝΙΚΩΝ ΔΟΜΩΝ (CATH και SCOP)
Στη συνέχεια θα έχουμε πρόσβαση σε δυο συστήματα κατηγοριοποίησης Πρωτεϊνικών Δομών. Η πρόσβασή μας στις καταχωρήσεις αυτές είναι δυνατή με τη βοήθεια του διαδικτύου, και τις ηλεκτρονικές διευθύνσεις
SCOP : http://scop.mrc-lmb.cam.ac.uk/scop/
CATH : http://www.biochem.ucl.ac.uk/bsm/cath/
Αρχικά, επιστέφουμε στην αρχική σελίδα αποτελεσμάτων του BLAST και από τις τέσσερις πρώτες ακολουθίες μας επιλέγουμε την ακολουθία εκείνη για την οποία υπάρχει πειραματικά προσδιοριζομένη δομή κατατεθειμένη στην PDB.
Αποθηκεύστε την καταχώρηση σας σε μορφή απλού κειμένου (Plain Text) στον κατάλογο 'C:\sequences\' με το όνομα 'agnosti.pdb'.
Ερώτηση:
Σημειώστε την συστηματική ταξινόμηση της πρωτεϊνικής δομής με βάση τα δυο συστήματα κατηγοριοποίησης CATH και SCOP. Παρατηρείται η ίδια ιεραρχική καταχώρηση ή όχι ;
Πόσα domains μπορούν να αναγνωρισθούν στην πρωτεϊνική δομή σας;
Δώστε λεπτομερή περιγραφή της δομής της πρωτεΐνης σας. Ποια είναι τα δευτεροταγή στοιχεία της και πως αυτά συνδέονται στο χώρο;
Αναγνωρίστε την παρουσία διαφόρων προσθετικών ομάδων ή άλλων υποκαταστατών στην δομή σας;
Σε συνδυασμό με τις πληροφορίες που σας παρέχουν άλλες βάσεις δεδομένων (ακολουθιών ή/και πρωτεϊνικών δομών) απαντήστε στην παρακάτω ερώτηση:
Πιστεύετε ότι η υψηλή ομολογία στο επίπεδο της αμινοξικής ακολουθίας μπορεί να χρησιμοποιηθεί για εξαγωγή δομικών-λειτουργικών συμπερασμάτων για μια άγνωστη ακολουθία; Δικαιολογήστε την απάντησή σας.
Ποια λειτουργία μπορείτε να αποδώσετε στην άγνωστη ακολουθία σας.
ΠΡΟΓΝΩΣΗ ΔΕΥΤΕΡΟΤΑΓΟΥΣ ΔΟΜΗΣ
Από τις διάφορες μεθόδους για την πρόγνωση δευτεροταγούς δομής που είναι διαθέσιμες, θα χρησιμοποιήσουμε τη μέθοδο 'SecStr', που έχει αναπτυχθεί στο εργαστήριό μας (Hamodrakas, 1988, Palaios and Hamodrakas, 1999).
Ερώτηση:
Συγκρίνετε τις προγνώσεις με την λυμένη δομή κατατεθειμένη στην PDB (C:\sequences\agnosti.pdb). Σχολιάστε τα αποτελέσματα.
ΣΥΜΠΕΡΑΣΜΑΤΑ
ser.glu.gln_ala.asn.ala.leu.tyr.ser.ile.ser|ile.ser|asx.glu.thr.thr.glu.arg|thr.his.ala.asn|thr.his.arg.glu.glu-asp|ala.asn.ala.leu.tyr.ser.ile.ser.
Μεταφράστε το παραπάνω κείμενο και απαντήστε αν συμφωνείτε ή όχι και γιατί με την πρόταση.
ΑΝΑΦΟΡΕΣ ΚΕΙΜΕΝΟΥ
Bairoch, A. and Apweiler, R. (1998) The SWISS-PROT protein sequence databank and its supplement TrEMBL in 1998, Nucleid Acids Res., 26, 38-42
Hubbard, T. J. P., Murzin, A. G., Brenner, S. E. and Chothia, C. (1997). SCOP: a structural classification of proteins database, Nucl. Acids Res., 25, 236-239
Murzin, A.G., Brenner, S.E., Hubbard, T. and Chothia, C.(1995) Scop: a structural classification of proteins database for the investigation of sequences and structures, Journal of Molecular Biology, 247 , 536-540
Orengo, C. A., Michie, A. D., Jones, D. T., Swindells, M. B. and Thornton, J. M. (1997) CATH - A hierarchic classification of protein domain structures, Structure, 5(8) , 1093-1108
Palaios, G.A., and Hamodrakas, S.J. (1999) SecStr: Secondary Structure prediction over the World Wide Web


